Illumina助力云南CDC破譯新型冠狀病毒全基因組序列
大道至簡,實干為要 | Illumina平臺助力云南省CDC快速破譯新型冠狀病毒全基因組序列
據云南廣播電視臺《都市條形碼》微信公眾號報道,1月21日,國家衛生健康委確認昆明市首例輸入性新型冠狀病毒感染的肺炎確診病例。云南省疾控中心急性傳染病防制所所長伏曉慶介紹,1月17日中午12點,實驗室收到患者采樣標本,下午3點就完成了核酸檢測,結果呈陽性。
1月18日凌晨1點,實驗室開始病毒全基因組序列測定,20日上午10點測序工作完成,經過與國家公布的新型冠狀病毒全基因組序列進行比對分析,證明測序結果與公布的序列高度同源,確證云南發現病例為新型冠狀病毒感染的肺炎確診病例。伏所長還表示,全基因組測序技術對于疫情研判、防控方案制定與措施采取,有至關重要的作用。

圖1:云南CDC技術專家正在進行病毒MiSeq測序前的準備(圖片來源:云南省CDC提供)

圖2:云南CDC技術專家正在生物安全柜中進行測序文庫樣品的前處理(圖片來源:云南省CDC提供)

圖3:利用Illumina平臺測定的、且在GenBank上公布的新冠病毒全基因組完整序列。(圖片來源:GenBank官方網站)
散彈槍法宏基因組測序(Shotgun metagenomics),即對待檢樣本的直接測序,專用于微生物群落的種屬鑒定及豐度測量。在傳染病領域,這一方法特別對新現病毒具有強大的檢出能力,規避了常規病毒分離培養過程可能引入的病毒變異,同時最大限度壓縮了病原檢出的時間。病毒是“一包基因”,基因組結構功能變異度高,沒有類似于細菌16S rRNA序列的分子標志物可供統一的進化親緣分析。
Jasper F. W. Chan et al. Clin. Microbiol. Rev. 2015; doi:10.1128/CMR.00102-14

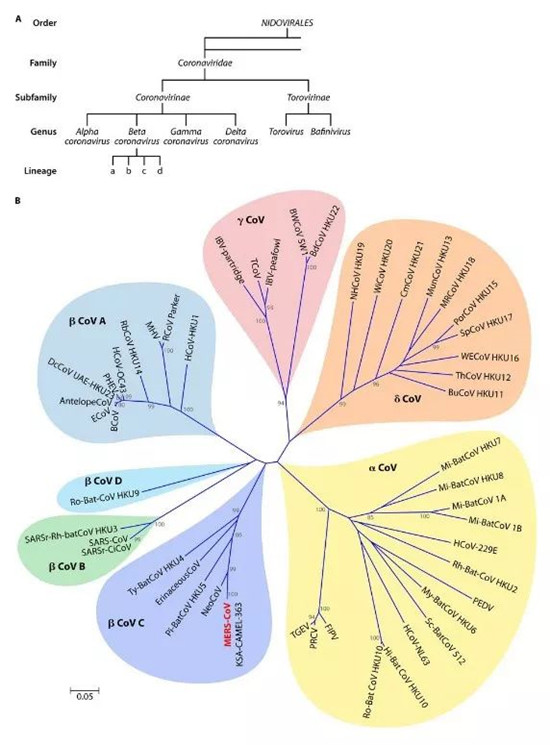
圖4:冠狀病毒科進化親緣關系
對于檢測諸如埃博拉、SARS冠狀病毒、MERS冠狀病毒,狂犬病毒等形形色色潛伏于環境或天然宿主中的RNA病毒而言,病毒本身滴度和背景核酸干擾是檢測成功與否的關鍵之一。核酸富集(Enrichment)與 病毒基因組互補DNA合成兩個技術環節顯得尤為重要。已發展出各式技術、方法用于提高核酸產量。
動物疫源性宿主(Zoonotic reservoirs)在傳播病原的過程中扮演著重要的角色。利用高通量測序能對動物宿主體內潛伏的病原進行廣譜篩查,已成為實現關口前移與治未病先進防疫理念倚仗的關鍵技術和重要手段。
以蝙蝠為例,所攜帶的病毒種群(組)存在天然的蓄存與流行。不同種類蝙蝠的病毒組有很大差異,其中就包含對脊椎動物具有致病性的病毒種屬,特別是高致病新現病毒,如甲型流感病毒、漢坦病毒、丙肝病毒、SARS樣beta群冠狀病毒、乳頭瘤病毒、細小RNA病毒等。
雖然本次武漢新冠病毒的來源、傳播途徑問題仍不清楚,但蝙蝠攜帶SARS樣冠狀病毒的發現早有報道,He等對268只蝙蝠肛拭子進行宏基因組測序,成功鑒定發現alpha、beta群冠病。其中于云南寶山檢出的SARS樣的冠狀病毒(LYRa11)基因組全序列顯示,它與人類SARS冠病序列一致率高達91%;尤其是刺突蛋白 人類細胞受體結合域(Receptor Binding domain,RBD)編碼序列與人SARS冠病高度近似,且能與SARS病人恢復期血清發生免疫反應,提示了兩者可能的重組親源關系。此外,我國科學家還從蝙蝠體內檢出MERS冠狀病毒的近親。
- 2025知楚全國總經銷商交流會在上海圓滿收官
- 易科泰多光譜熒光成像系統在海南熱帶海洋學院安裝
- CytScop®Mini智能細胞計數儀亮相斯坦福大學
- 熱烈祝賀易科泰榮膺2025全球數字農業品牌企業
- 湖南赫西參加"儀器維保聯盟"啟動儀式,共繪美好藍圖
- 飛納電鏡用戶培訓會廈門站圓滿落幕
- ARveo8顯微鏡助力首例頸深LVA術成功逆轉AD認知衰退
- 熱烈慶祝西美杰正式成為 Virusys 品牌代理商!
- 普瑞麥迪成為瑞士3Brain中國區戰略合作伙伴
- 2024年"MCE中國生命科學研究促進獎"評獎結果公布
- 全球視野,本土深耕-丹納赫中國生命科學平臺戰略升級
- 安捷倫向哥倫比亞大學Pawel Muranski博士授予ARC獎
- INTEGRA WELLJET試劑分液器現已支持SiLA-2集成
- 安捷倫xCELLigence實時細胞分析助力Autolus獲批
- 易科泰植物葉綠素熒光成像系統交付西安理工大學
- 森西賽智聯合Vizgen在兩場大會正式推出MERFISH2.0
- 怡美通德邀您參加第三屆國際單細胞及空間組學大會
- 10x Genomics技術方案更新(2月),新手冊等您獲取
- 2025中國細胞與基因治療大會第五屆年會招展通知
- 10x和CZI及Ultima Genomics合作啟動十億細胞項目
- 第二屆中國國際生殖醫學健康大會通知
- 森西賽智聯合Vizgen發布MERFISH保存樣本空轉方案
- 第五屆多組學科研與臨床應用大會(北京站)通知
- 2025年10x Genomics校園行邀請—北京中醫藥大學站
- 華大智造發布桌面式測序儀T1+,24小時可完成Tb級測序
- 因美納推出多組學創新方案,解鎖更深入的生物學見解
- 因美納推出全新空間轉錄組學技術
- 真邁生物再次獲批“十四五”國家重點研發計劃
- 因美納被列入不可靠實體清單,國產替代,誰是最優解
- IGC 2025第九屆免疫基因及細胞治療大會通知
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com






