PEAKS中的蛋白質翻譯后修飾分析強大功能詳解
蛋白質翻譯后修飾(PTM),如磷酸化、泛素化、乙酰化和甲基化,在信號傳導和調控過程、蛋白質活性和降解、基因表達調控等多種生物過程中起著關鍵作用。PTM的鑒定和表征對于全面了解細胞生物學和人類疾病至關重要,并且具有廣泛的應用。由于蛋白質數據庫中PTM信息通常不存在或不完整,因此通過質譜法鑒定PTM給傳統的數據庫搜索方法帶來了許多挑戰。
PEAKS中包含的高級算法可最大限度地提高PTM鑒定和PTM分析。PEAKS PTM的翻譯后修飾鑒定是通過整合PEAKS數據庫搜索和De Novo測序結果來實現的。PTM Profile工具通過提供定性和定量信息以及對結果的可視化和直接導出,進一步協助您的PTM研究。
PEAKS中的PTM分析
PEAKS中的PTM分析
- 通過PEAKS數據庫搜索來鑒定PTM
- 通過PEAKS PTM發現非指定的或隱藏的修飾來達到最大程度的鑒定PTM
- 通過PEAKS PTM對PTM進行定量分析

通過LC-MS/MS進行翻譯后修飾(PTM)分析

通過LC-MS/MS進行翻譯后修飾(PTM)分析
PTM 鑒定
PTM鑒定工作可以通過PEAKS數據庫搜索和PEAKS PTM搜索來完成。
01通過數據庫搜索
在數據搜索的方法中,僅僅對于有限個數的一些常見翻譯后修飾可以被定義為可變修飾。
蛋白的結果視圖中,鑒定到的支持性多肽證據以藍色的條,映射到選中的蛋白的序列上。通過點擊多肽,該多肽的二級質譜圖將會顯示出來,您可以很容易地檢查質譜數據的注釋。
在二級譜圖的窗口中顯示的多肽序列里,帶有修飾的氨基酸殘基以小寫字母表示,例如在下圖中,序列DTLmISR的甲硫氨酸。
蛋白的結果視圖中,鑒定到的支持性多肽證據以藍色的條,映射到選中的蛋白的序列上。通過點擊多肽,該多肽的二級質譜圖將會顯示出來,您可以很容易地檢查質譜數據的注釋。
在二級譜圖的窗口中顯示的多肽序列里,帶有修飾的氨基酸殘基以小寫字母表示,例如在下圖中,序列DTLmISR的甲硫氨酸。

02通過PEAKS PTM 搜索
PEAKS PTM是整合了強大的de novo算法和數據庫搜索算法的,特別為發現隱藏修飾而設計的功能模塊。
這種多輪搜索方法在數據分析工作流圖所描述:
01.對每一張譜圖進行de novo測序
02.PEAKS數據庫(DB)算法用于蛋白質鑒定。在這一輪中可以指定一些高頻的常見PTM,以更好地提高靈敏度
03. PEAKS的PTM算法用于鑒定更多的翻譯后修飾。在這一輪,僅僅通過de novo高置信度打分,并且同時沒有在數據庫中匹配的譜圖被映射到已鑒定的蛋白上。用戶可以定義任意多個的PTM,或者他們可以簡單的運行一輪PTM搜索,可以實現650種Unimod數據庫中翻譯后修飾的同時檢索。
PEAKS PTM模塊鑒定到另外的修飾,例如下圖,除了在數據搜索得到的,在DTLMISR序列中甲硫氨酸的oxidation(圖中黑色)以外,還有一種修飾dethiomethyl(圖中紅色指向)。
這種多輪搜索方法在數據分析工作流圖所描述:
01.對每一張譜圖進行de novo測序
02.PEAKS數據庫(DB)算法用于蛋白質鑒定。在這一輪中可以指定一些高頻的常見PTM,以更好地提高靈敏度
03. PEAKS的PTM算法用于鑒定更多的翻譯后修飾。在這一輪,僅僅通過de novo高置信度打分,并且同時沒有在數據庫中匹配的譜圖被映射到已鑒定的蛋白上。用戶可以定義任意多個的PTM,或者他們可以簡單的運行一輪PTM搜索,可以實現650種Unimod數據庫中翻譯后修飾的同時檢索。
PEAKS PTM模塊鑒定到另外的修飾,例如下圖,除了在數據搜索得到的,在DTLMISR序列中甲硫氨酸的oxidation(圖中黑色)以外,還有一種修飾dethiomethyl(圖中紅色指向)。


PTM 位點確證
修飾的確切位置可由譜圖中存在決定位點的碎片離子來確定。在下圖所示的例子中,MS2譜圖中b-11和b-12離子的存在決定了天冬酰胺在第12位發生脫氨基。
PEAKS為用戶提供兩個選擇以確定可信的修飾位點:
01.最小離子強度,這要求MS/MS譜圖中決定位置的碎片離子的相對強度必須高于用戶輸入的數值。
02.Ascore,將歧義分數計算為 -10 × log10 P。p 值表示肽偶然匹配的可能性。因此,Ascore越高置信度越高。
這兩種方法都提供了PTM修飾位點定位的置信度。如果達到了用戶選擇方法的閾值,則PTM將顯示在蛋白質覆蓋率視圖中殘基上方的彩色框中。
PEAKS為用戶提供兩個選擇以確定可信的修飾位點:
01.最小離子強度,這要求MS/MS譜圖中決定位置的碎片離子的相對強度必須高于用戶輸入的數值。
02.Ascore,將歧義分數計算為 -10 × log10 P。p 值表示肽偶然匹配的可能性。因此,Ascore越高置信度越高。
這兩種方法都提供了PTM修飾位點定位的置信度。如果達到了用戶選擇方法的閾值,則PTM將顯示在蛋白質覆蓋率視圖中殘基上方的彩色框中。

PTM 定量分析
PEAKS PTM Profile提供了對定量信息直接地可視化和總結(例如,在鑒定到的位點,修飾和未修飾形式的豐度)。
PEAKS PTM Profile提供了對定量信息直接地可視化和總結(例如,在鑒定到的位點,修飾和未修飾形式的豐度)。
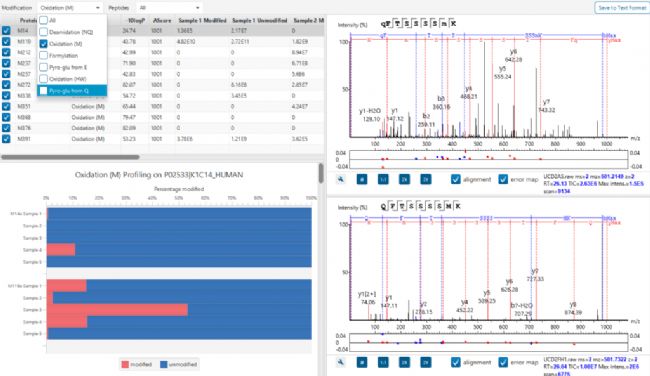
PTM profile的結果可以導出圖像和包含有詳細定量信息的CSV格式結果。


Reference
Han, X. et al. PEAKS PTM: Mass Spectrometry Based Identification of Peptides with
Unspecified Modifications. Journal of Proteomics Research. 10(7), 29302936. 24/05
/2011.
Unspecified Modifications. Journal of Proteomics Research. 10(7), 29302936. 24/05
/2011.
如果您想深入了解更多關于PEAKS的PTM分析,歡迎掃描下方二維碼關注我們!

Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com





