深藍云數字PCR在檢測廢水中SARS-CoV- 2再生數評估Rww的應用
法國Stilla Technologies公司的naica®微滴芯片數字PCR系統用于評估有效再生數Re(effective reproduction number)。
有效再生數Re(effective reproduction number)為一段時間內由單個感染者引起的新感染的平均數量,作為監測疾病動態、是區域和國家政策提供信息以及評估干預措施有效性的一個關鍵指標,在COVID-19大流行期間被廣泛用于跟蹤疾病動態。通常有效再生數Re根據臨床病例數據進行評估(以下簡稱Rcc),如觀察病例、住院和/或死亡,可能因檢測和報告的變化而產生偏差。瑞士聯邦理工學院科學家在medrxiv發布的跟蹤研究表明,廢水中SARS-CoV-2 RNA的動態可以用于近實時評估Re,不依賴于臨床數據,也不存在來自臨床測試和報告策略的相關偏差。

文章中,使用數字PCR方法的定量檢測廢水和固沉積物病毒RNA載量,提供一個獨立的數據集來估計Re(以下稱為Rww),能對原有Rcc評估補充,以提供一個更完整的圖像式傳染動力學。廢水中SARS-CoV-2 RNA的縱向測量可用于評估不同地區的Re,并提供對COVID-19動態的獨立評估。鑒于在此次大流行期間廣泛進行廢水取樣,這些Re值可直接作為一種快速、低成本的方法,為公共衛生政策提供信息。該方法可適用于其他病原體,包括那些沒有臨床數據的病原體。
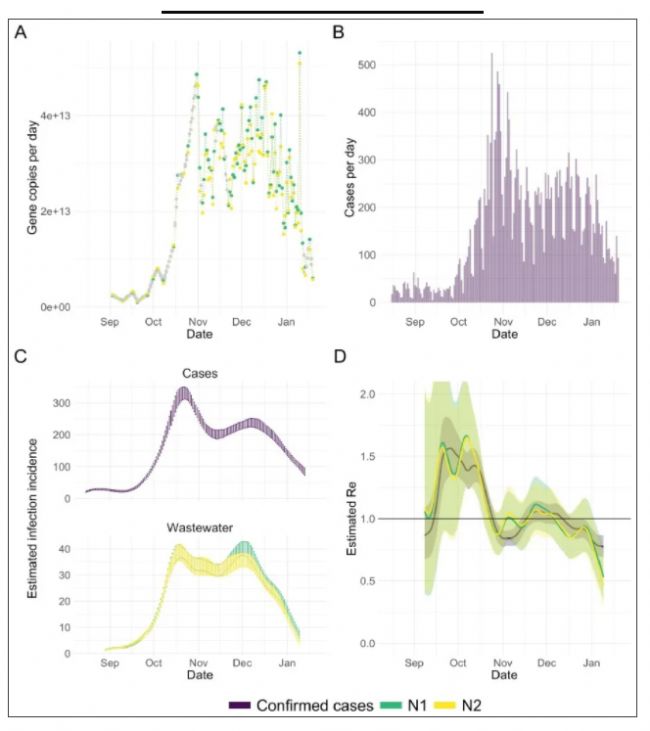
圖1:蘇黎世(CHE)廢水Rww
評估:A. 2020年9月1日至2021年1月20日期間測定的N1和N2標記(分別為綠色和黃色)的RNA載量。B. 同一時期確診病例(紫色)。C. 根據病例報告得到的感染發生率時間序列(上)和根據N1和N2標記物的歸一化RNA載量(下),分別為綠色和黃色。歸一化方法請見原文中方法描述,條形區域表示50次重復的平均值±標準偏差。D. Rww與Rcc 對比。彩色線表示原始數據點,帶狀區表示50次重復的的置信度95%的置信區間。
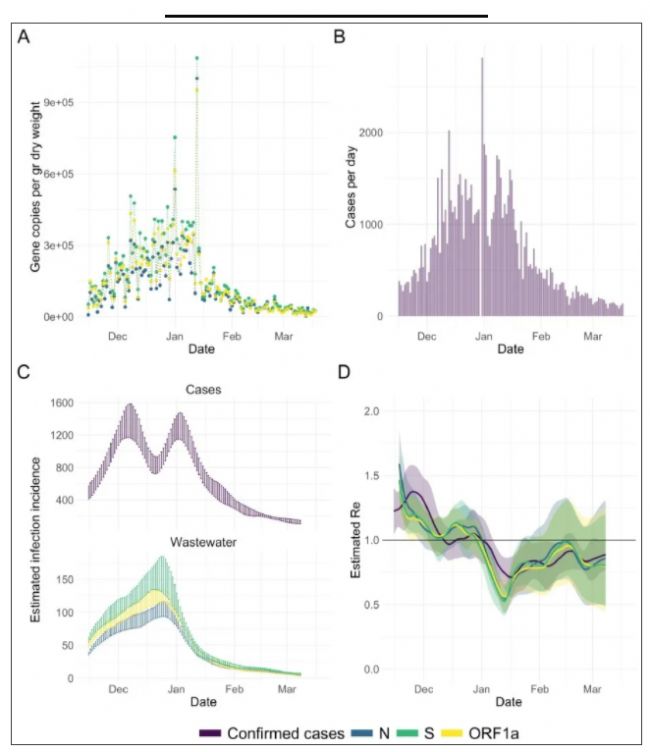
圖2:圣何塞(美國)污泥Rww 評估:A. 2020年11月15日至2021年3月19日期間測定的
N、S和ORF1a基因(分別為藍色、綠色和黃色)RNA載量。B. 同期確診病例(紫色)。
C. 根據病例報告的感染發生率時間序列(上)和歸一化的RNA載量(下)。歸一化方法請參考原文方法。條形圖表示50次重復平均值±標準偏差。(D)Rww 與Rcc 對比。彩色線表示原始數據點,帶狀區表示50次重復置信度95%的置信區間。
文章發現Rww 與Rcc結果一致,但兩者時間上存在滯后,圖2D。通常來說,Rww 或者Rcc 時間不準確的原因,推測是因為未能在最佳時間捕捉脫落,或者因為在觀察期間臨床病例的報告延遲改變。在這種情況下,Rcc可能由于漏報和增加測試延遲,導致存在偏差。根據測試調整的案例重新估計,結果更接近于Rww,尤其是11月/ 12月,圖S1B。
文章中作者調整了采樣頻率和易感性,通過建立噪聲模型和對脫落負荷分布(SLD)進行反卷積,將廢水中的RNA測量與原始感染發生率聯系起來,準確描述脫落的時間動態和足夠大的集水區,個體差異可能會趨于平均,隨著前瞻性抽樣研究報告結果,可以得出最優SLD ,從而提供更準確的廢水和固體沉積物的Rww。
從廢水中獲得Re為追蹤疾病動態提供了一種獨立的方法。基于廢水的流行病學檢測方法可在全球用于跟蹤COVID-19大流行(https://www.covid19wbec.org/covidpoops19),Rww 評估方法非常強大,不受測試和報告策略的影響,因此跨地理區域的評估更具可比性。通過估計Rww進行廢水監測是一種低成本、快速并可對不同區域水質進行比較的方法,同時也能實現以接近實時的方式跟蹤疾病傳播動態。
medRxiv是由美國冷泉港、BMJ和耶魯大學聯合運營的免費的、非營利性服務平臺。2019年6月25日正式上線,專注于臨床研究的科研人員可以在medRxiv平臺上分享未經過同行評議的研究,從而有助于及時傳播最新的發現,也有助于研究人員接收反饋進而完善研究內容。
原文鏈接:https://doi.org/10.1101/2021.04.29.21255961
針對于naica®微滴芯片數字PCR系統,我們提供新型冠狀病毒(2019-nCoV)核酸檢測試劑盒,貨號:AK900103。參考國家CDC與WHO指南方法,生信分析新型冠狀病毒的開放讀碼框1ab(open reading frame, ORF1ab)、核殼蛋白(nucleoprotein,N)區域保守序列,優化引物探針,降低病毒變異的影響;利用三熒光通道的優勢,加入人源管家基因作為內標進行監控,實現單孔多重三基因檢測,結果更可靠。詳情點擊:https://mp.weixin.qq.com/s/uXv2JLTg1YJsQJFW9IGHzA

naica®微滴芯片數字PCR系統
法國Stilla Technologies公司的naica®微滴芯片數字PCR系統在進行核酸檢測時具有獨特的優勢。該系統利用cutting-edge微流體創新型芯片—Sapphire芯片(或高通量Opal芯片)作為數字PCR過程的耗材。樣品通過毛細通道網格以30,000個微滴的形式進入2D芯片中。3色熒光檢測儀器,整個流程只需要2.5小時,并可進行數據的質控和結果追溯分析,獲得的數據真實可靠。
naica®六通道微滴芯片數字PCR系統
法國Stilla Technologies公司naica®六通道微滴芯片數字PCR系統,源于Crystal微滴芯片數字PCR技術,自動化微滴生成和擴增,每個樣本孔可實現6熒光通道的檢測,智能化識別微滴并進行質控,3小時內即可獲得至少6個靶標基因的絕對拷貝數濃度。






