不依賴泛素蛋白酶降解途徑的新型 PROTAC 應用
前文我們已經介紹過,PROTAC 分子的設計之初,靶蛋白配體、E3 連接酶配體和兩者中間的連接子三者應該分別考量。
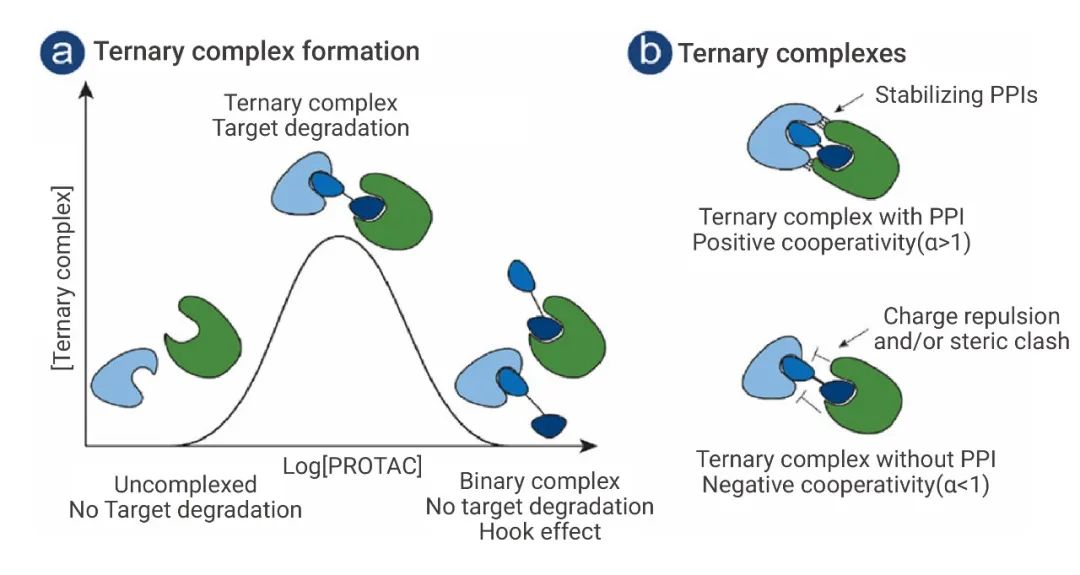
在 PROTAC 介導的蛋白降解途徑中,其中靶標配體 (POI)-PROTAC-E3 連接酶等三元復合物的有效形成同樣起到重要作用。三元復合物的任一單元的濃度改變帶來的不僅僅是線性變化,“鐘” 形模型常常被用來解釋這樣的活性與濃度相關性,文獻中提及的 hook effect 也正是形容這種情形。
另外,在活性研究中,協同效應 (cooperativity, α) 這一詞匯,被用于解釋 PROTAC 受 POI 與 E3 連接酶配體間蛋白蛋白相互作用 (PPI) 的影響。當 α 大于 1 時,POI 和 E3 是正協同效應,對三元復合物的形成有利。
圖 1. PROTAC 降解能力的“鐘”形模型與協同效應示意圖[1]
不依賴泛素蛋白酶的降解體系
1. 依賴溶酶體途徑的降解類分子
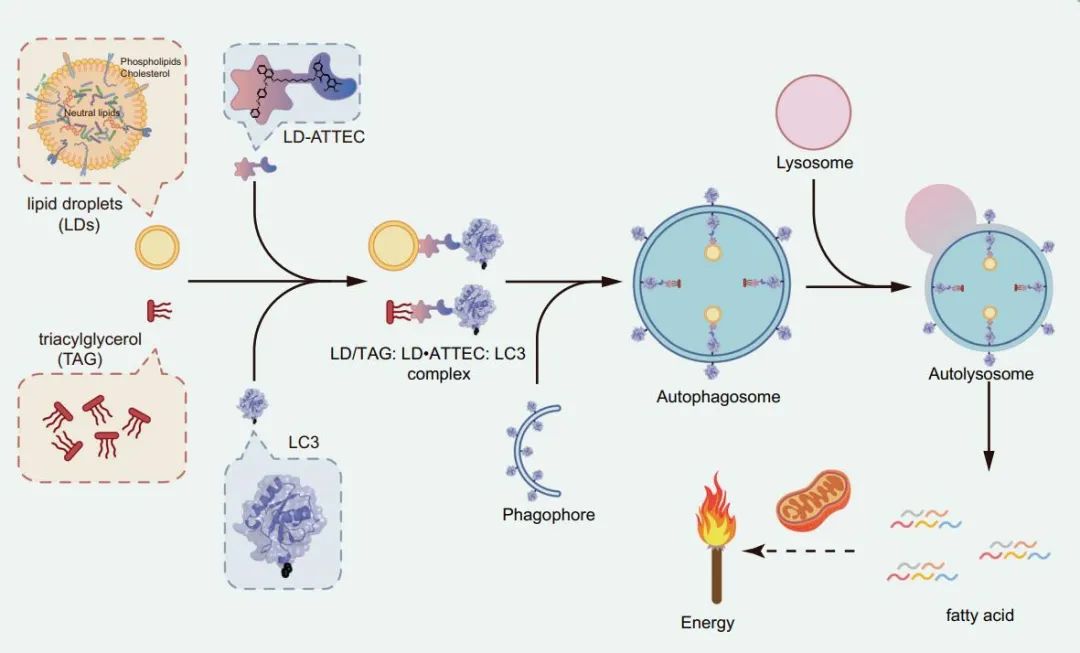
PROTAC 主要依賴泛素-蛋白酶體途徑,其對應降解的大多為胞質蛋白與核蛋白。Bertozzi 教授的研究團隊報道了一項靶向胞外蛋白的降解技術——溶酶體靶向嵌合體 (LYTAC)。已證明 LYTAC 成功降解了表皮生長因子受體 (EGFR)、程序性死亡配體 1 (PD-L1) 等膜上蛋白。對于細胞中一些非蛋白成分,Targeting lipid droplets for autophagic degradation by ATTEC 一文展示了一種通過自噬束縛化合物 (ATTECs) 降解非蛋白質生物分子的新策略,使用脂滴作為示例靶標并成功降解。
LD·ATTECs 通過疏水作用與 LD 以及自噬體蛋白 LC3 結合,形成LD/TAG LD·ATTEC LC3三元復合物,復合物與自噬體溶酶體融合并最終被自噬體降解。
但不論是 PROTAC 還是上述其他方式,它們都靶向真核生物細胞內物質的降解,均不能在細菌和其他原核生物中發揮功能。
2. 依賴 ClpCP 酶的 BacPROTAC
今年 6 月,Morreale 團隊等人在 Cell 發表的 BacPROTACs mediate targeted protein degradation in bacteria 一文報道了一種用于革蘭氏陽性菌和分枝桿菌靶向蛋白降解的創新技術,BacPROTAC。
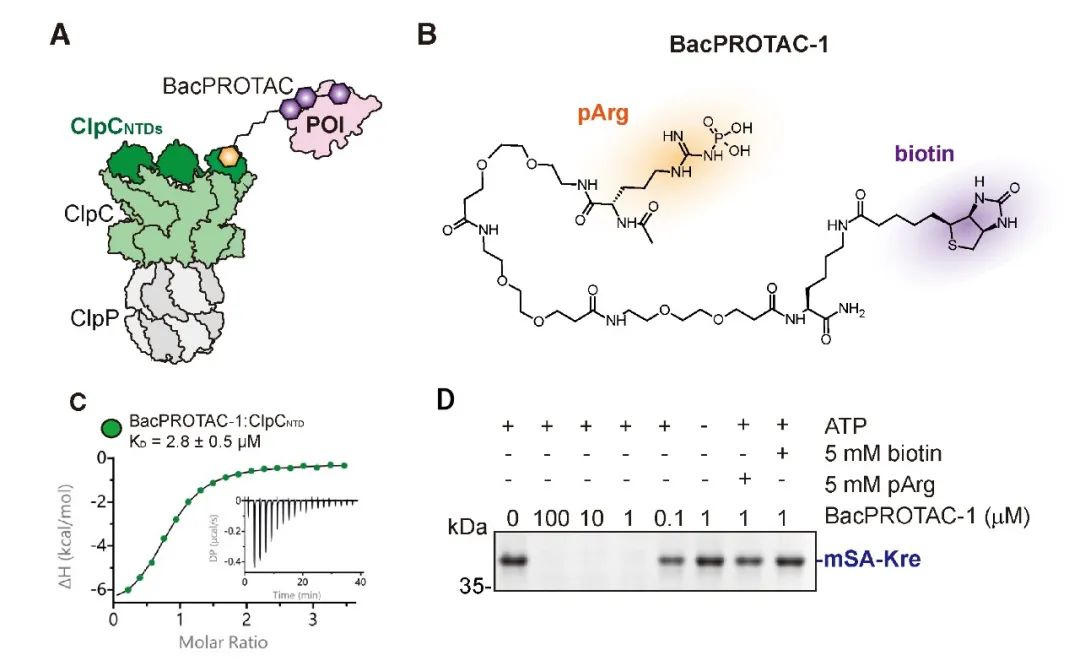
他們基于 Clausen 實驗室之前的一項發現:在枯草芽孢桿菌和其他革蘭氏陽性菌中,AAA 去折疊酶 ClpC 和蛋白酶 ClpP 組成的 ClpC–ClpP (ClpCP) 蛋白酶是降解細菌中未折疊和聚集蛋白質的重要蛋白水解酶。由于磷酸精氨酸的對接位點位于 ClpC ATP 酶的氨基末端結構域,磷酸精氨酸 (pArg) 可作為 ClpCP 蛋白酶復合體的降解標簽。
Morreale 等人報道的細菌 PROTACs (BacPROTACs) 由 POI 配體、Linker 和 ClpCNTDs 配體組成,可誘導細菌這種沒有泛素蛋白酶體的非真核生物蛋白的體內外降解。如圖 3A,研究團隊在體外首先以單體鏈霉親和素 (mSA) 作為模型蛋白,通過 BacPROTAC 將 pArg (ClpCNTD 配體) 與生物素 (mSA 的配體) 結合,形成 BacPROTAC-1 三元復合物,有效降解靶蛋白。BacPROTAC-1 與 mSA 和 ClpCNTD 結合的 KD 為 3.9 和 2.8 μM (如圖 3C)。
圖 3. BacPROTAC-1 對枯草芽孢桿菌 ClpCP 的體外重編程[6]
由于磷精氨酸的化學穩定性等方面存在問題,在體內的應用有限。作者團隊將內源性的磷酸化精氨酸殘基 (pArg) 換成了高選擇性的 sCym-1。sCym-1 不但可以和枯草芽孢桿菌的 ClpCP 結合,還可以與分歧桿菌的 ClpC1P1P2 結合。
由于細胞生物素會競爭性與 mSA 結合,一定程度上阻礙 BacPROTAC 活性 (圖 5),作者團隊確定以 Bromodomain-1 (BD1) 作為更吸引人的模型底物,并將 JQ1 作為新的POI配體,天然環素 dCymM 作為 ClpCNTD 結合基團。BacPROTACs 以濃度依賴性的方式促進 BRDTBD1 的降解。
圖 5. BacPROTACs 可以對分枝桿菌 ClpC1P1P2 進行重編程[6]
上:BRDTBD1-directed BacPROTACs: 通過不同的 Linker 和連接位點連接 JQ1 到dCymM。下:分枝桿菌 ClpC1P1P2 與 BD1 孵育后降解的 SDS-PAGE 分析。
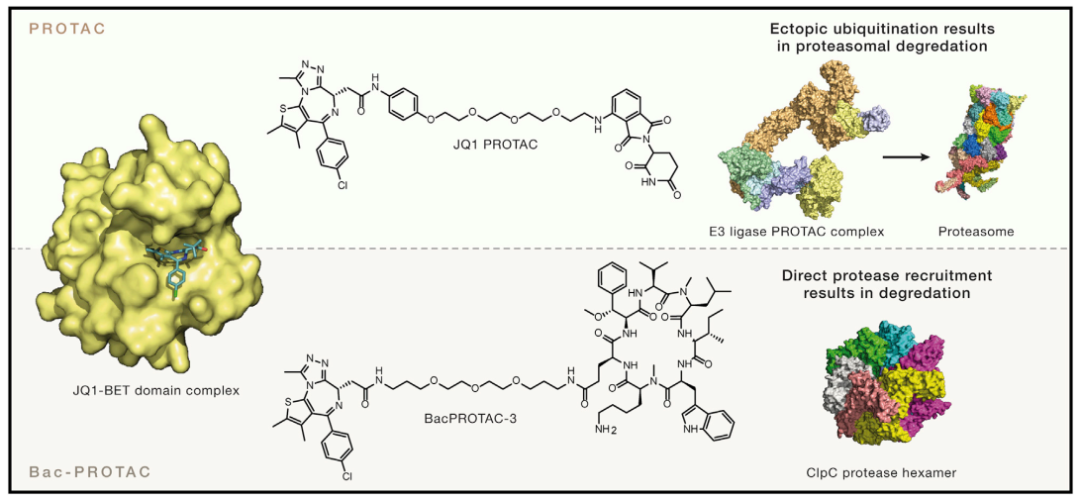
總之,如圖 6 所示:PROTACs 通過招募 E3 連接酶來誘導蛋白質降解,E3 連接酶使得靶蛋白貼上泛素化標簽,最后蛋白酶體降解泛素化的蛋白質。
BacPROTACs 以高度特異的方式將細菌 ClpCP 蛋白酶定向到底物上,它不但可以誘導底物和蛋白酶之間的接近,并且通過結合誘導無活性的 ClpCP 十聚體重組為有活性的六聚體形式,直接啟動 ClpCP 水解靶標蛋白。
圖 6. 蛋白質降解的異雙功能方法[5]
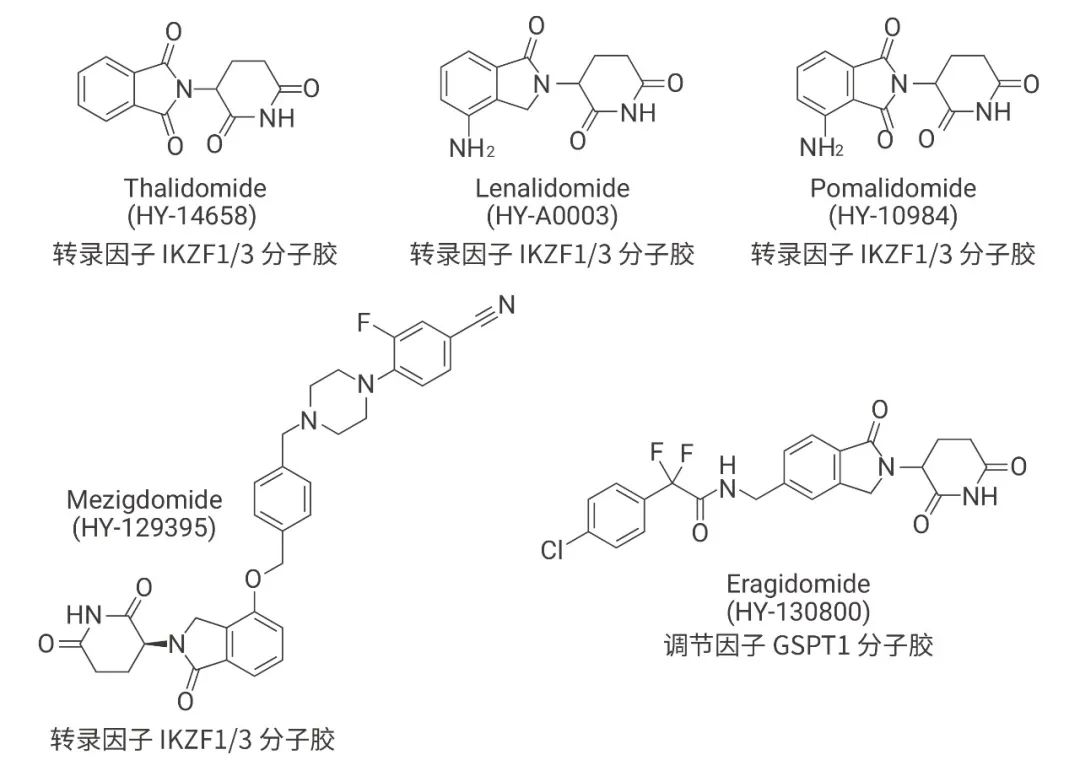
在降解劑分類里,分子膠 (Molecular glues) 指同樣發揮誘導蛋白降解作用卻無 Linker 連接的一類分子,如Thalidomide、CC-92480、CC-90009 等結構。分子膠在結構上更接近傳統小分子,在膜透過性和生物利用度上更具優勢,是 PROTAC 型降解劑的一個改造方向。
降解能力評價
降解劑的最終目的是降解相關的蛋白, 無論是依賴泛素酶途徑的 PROTAC,依賴蛋白酶體途徑的 LYTAC,依賴 (ClpCP) 蛋白酶的 BacPROTAC。除 Kd 值測定之外,Western Blot 實驗在驗證蛋白降解方面更為直觀,因此常常被用于 PROTAC 類分子活性的測試。
總結
PROTAC 已經衍生出一類 “POI 配體-Linker-降解系統導向物” 模式的分子,這類分子補足了 PROTAC 作為降解劑在某些蛋白與非蛋白分子降解上的不足,賦予了降解劑這一概念更多的可能性。除了分子自身作用機制帶來活性測試結果上與常規分子的差異,合適的降解途徑對活性的影響尤其巨大。
MCE 是全球前沿的科研化學品和生物活性化合物供應商,可以為科學家提供 PROTAC 類相關產品,目前我們已有 PROTAC、AUTAC、ATTEC、分子膠以及它們的構成模塊等各種產品在線。同時,我們還提供 PROTAC 類產品的一體化合成服務。
MCE 的所有產品僅用作科學研究或藥證申報,我們不為任何個人用途提供產品和服務。
參考文獻
1. Jin J, Wu Y, Chen J, Shen Y, Zhang L, Zhang H, Chen L, Yuan H, Chen H, Zhang W, Luan X. The peptide PROTAC modality: a novel strategy for targeted protein ubiquitination. Theranostics. 2020 Aug 8;10(22):10141-10153.
2. Fischer F, Alves Avelar LA, Murray L, Kurz T. Designing HDAC-PROTACs: lessons learned so far. Future Med Chem. 2022 Jan;14(3):143-166.
3. Pettersson M, Crews CM. PROteolysis TArgeting Chimeras (PROTACs) - Past, present and future. Drug Discov Today Technol. 2019 Apr;31:15-27.
4. Ahn G, Banik SM, Miller CL, Riley NM, Cochran JR, Bertozzi CR. LYTACs that engage the asialoglycoprotein receptor for targeted protein degradation. Nat Chem Biol. 2021 Sep;17(9):937-946.
5. Fu Y, Lu B. Targeting lipid droplets for autophagic degradation by ATTEC. Autophagy. 2021 Dec;17(12):4486-4488.
6. Morreale FE, Kleine S, Leodolter J, Junker S, Hoi DM, Ovchinnikov S, Okun A, Kley J, Kurzbauer R, Junk L, Guha S, Podlesainski D, Kazmaier U, Boehmelt G, Weinstabl H, Rumpel K, Schmiedel VM, Hartl M, Haselbach D, Meinhart A, Kaiser M, Clausen T. BacPROTACs mediate targeted protein degradation in bacteria. Cell. 2022 Jun 1:S0092-8674(22)00593-1.
7. Schreiber SL. The Rise of Molecular Glues. Cell. 2021 Jan; 184(1): 3-9.
8. Li Z, Wang C, Wang Z, Zhu C, Li J, Sha T, Ma L, Gao C, Yang Y, Sun Y, Wang J, Sun X, Lu C, Difiglia M, Mei Y, Ding C, Luo S, Dang Y, Ding Y, Fei Y, Lu B. Allele-selective lowering of mutant HTT protein by HTT-LC3 linker compounds. Nature. 2019 Nov;575(7781):203-209.
9. Grohmann, C., Magtoto, C.M., Walker, J.R. et al. Development of NanoLuc-targeting protein degraders and a universal reporter system to benchmark tag-targeted degradation platforms. Nat Commun. 2022 Apr; 13:2073.