
|
Arraystar m6A-mRNA&lncRNA表觀轉(zhuǎn)錄組芯片,可定量檢測mRNA&lncRNA中m6A的表觀轉(zhuǎn)錄修飾水平。
|
|||||||||||||||||
| [發(fā)表評論] [本類其他服務(wù)] [本類其他服務(wù)商] | ||||||||||||||||||
| 服務(wù)商: 上海康成生物工程有限公司 | 查看該公司所有服務(wù) >> |
Arraystar m6A-mRNA&lncRNA表觀轉(zhuǎn)錄組芯片,可定量檢測mRNA&lncRNA中m6A的表觀轉(zhuǎn)錄修飾水平。
芯片優(yōu)勢
與MeRIP-seq相比,Arraystar表觀轉(zhuǎn)錄組芯片有其獨(dú)特的優(yōu)勢:
• 可同時(shí)檢測何種轉(zhuǎn)錄本攜帶修飾,以及不同條件下的修飾差異,更重要的是,還可以檢測每一種轉(zhuǎn)錄本的修飾比例
• 全面覆蓋了編碼基因和非編碼基因,甚至用MeRIP-seq方法很難檢測的lncRNA也可用芯片檢測
• 不需要去除rRNA,比MeRIP-seq更簡單便捷
• 樣本需求量少,總RNA量低至1ug
• 適用于多種樣本,比如降解的FFPE樣本,血清/血漿/全血樣本
Arraystar mRNA&lncRNA表觀轉(zhuǎn)錄組芯片產(chǎn)品列表
| 服務(wù) | 芯片 | 表觀修飾 | 規(guī)格 | 描述 |
|---|---|---|---|---|
| Human mRNA&lncRNA 表觀轉(zhuǎn)錄組芯片技術(shù)服務(wù) | Human mRNA&lncRNA 表觀轉(zhuǎn)錄組芯片 | m6A | 8×60K | 44,122 mRNAs; 12,496 lncRNAs; 3,813 Mid-size ncRNAs |
| Mouse mRNA&lncRNA 表觀轉(zhuǎn)錄組芯片技術(shù)服務(wù) | Mouse mRNA&lncRNA 表觀轉(zhuǎn)錄組芯片 | m6A | 8×60K | 48,161 mRNAs; 8,393 lncRNAs; 4,087 Mid-size ncRNAs |
| Rat mRNA&lncRNA 表觀轉(zhuǎn)錄組芯片技術(shù)服務(wù) | Rat mRNA&lncRNA 表觀轉(zhuǎn)錄組芯片 | m6A | 4*44k | 27,770 mRNAs; 10,582 lncRNAs; 2,505 Mid-size ncRNA |
芯片特點(diǎn)
芯片可定量每一種轉(zhuǎn)錄本的修飾百分比
同一種RNA轉(zhuǎn)錄本的修飾亞群和非修飾亞群,由于其結(jié)構(gòu)和結(jié)合蛋白的不同,會(huì)導(dǎo)致不同的命運(yùn)(圖1)。重要的是,轉(zhuǎn)錄本的修飾比例與它們的功能高度相關(guān)。然而目前的修飾檢測方法,比如MeRIP-seq(m6A-seq),可以確定修飾的位置,卻無法對一個(gè)特定RNA轉(zhuǎn)錄本修飾和非修飾部分的相對含量進(jìn)行定量。Arraystar表觀轉(zhuǎn)錄組芯片能夠在同一個(gè)芯片中通過雙熒光通道的方法檢測每個(gè)轉(zhuǎn)錄本修飾亞群和非修飾亞群的百分比(圖 2),同時(shí)對何種轉(zhuǎn)錄本發(fā)生修飾和不同條件下轉(zhuǎn)錄本的修飾差異進(jìn)行鑒定。
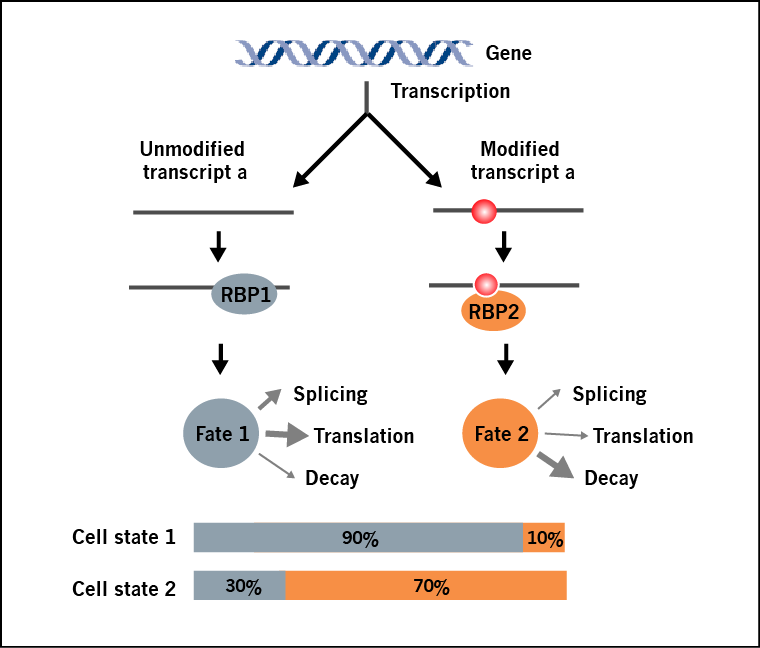
圖 1.同一種RNA轉(zhuǎn)錄本,隨著其修飾的化學(xué)計(jì)量數(shù)發(fā)生變化,其功能也隨之改變。在某一種細(xì)胞條件下,攜帶修飾的“RNA 轉(zhuǎn)錄本a”所占百分比可能非常低(比如,細(xì)胞狀態(tài)1),但在另外一種條件下豐度變的很高(比如,細(xì)胞狀態(tài)2)。通過引起RNA結(jié)構(gòu)改變,或招募修飾閱讀蛋白, “轉(zhuǎn)錄本a”的命運(yùn)發(fā)生變化,比如從蛋白翻譯轉(zhuǎn)變?yōu)镽NA降解。
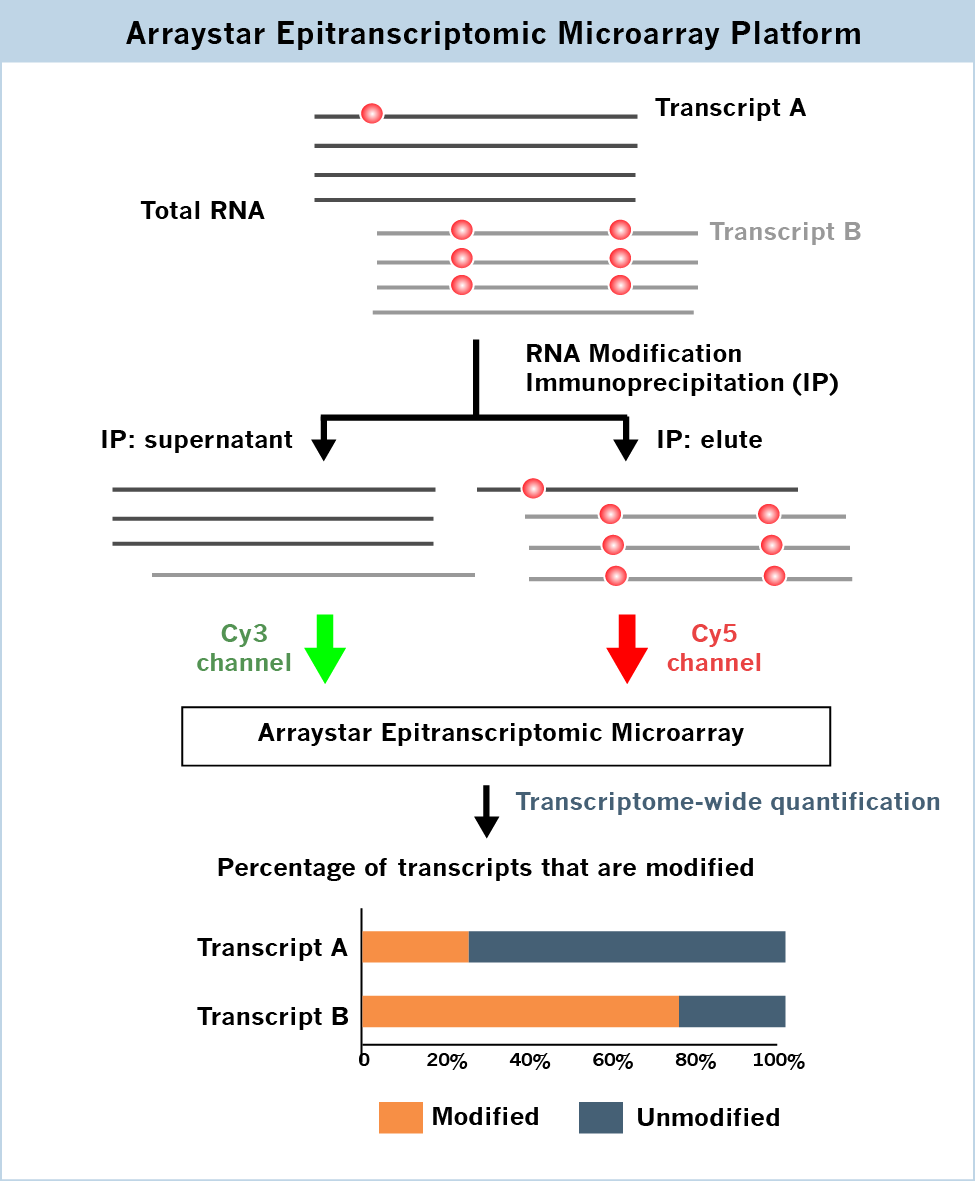
圖 2.Arraystar表觀轉(zhuǎn)錄組芯片可以在同一張芯片上使用Cy5檢測免疫共沉淀的修飾RNA,用Cy3檢測上清中的非修飾RNA,從而檢測每一個(gè)轉(zhuǎn)錄本中修飾和非修飾的百分比。選擇性剪切產(chǎn)生的轉(zhuǎn)錄本異構(gòu)體可以被轉(zhuǎn)錄本特異性探針?biāo)鶛z測。
覆蓋編碼基因和非編碼基因的表觀轉(zhuǎn)錄組
•Arraystat mRNA&lncRNA Epitranscriptomic Microarrays
適用于mRNA, lncRNA, pre-miRNA, pri-miRNA, snoRNA, 和 snRNA
•Arraystar circRNA Epitranscriptomic Microarray
適用于環(huán)狀RNA,芯片收集了高可信度的環(huán)狀RNA(在≥2個(gè)實(shí)驗(yàn)組和≥4個(gè)樣本中有表達(dá))
•對MeRIP-seq很難檢測的RNAs(比如lncRNA和circRNA),也具有高靈敏度和準(zhǔn)確度
轉(zhuǎn)錄本特異性的RNA修飾檢測
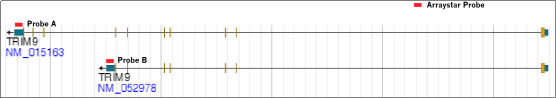
選擇性剪切產(chǎn)生的轉(zhuǎn)錄本異構(gòu)體具有組織特異性和不同的生物功能。例如,TRIM9短的異構(gòu)體(NM_052978),而非長的異構(gòu)體(NM_015163),能夠促進(jìn)DNA和RNA病毒引起的I型干擾素的表達(dá)。轉(zhuǎn)錄本異構(gòu)體的修飾百分比發(fā)生改變,常與生物功能和疾病相關(guān)。
Arraystar表觀轉(zhuǎn)錄組芯片,使用外顯子和跨剪接位點(diǎn)特異性探針,確保了每個(gè)轉(zhuǎn)錄本異構(gòu)體修飾水平檢測的可信度與精確性,提供了更深層次的表觀轉(zhuǎn)錄組學(xué)信息(圖 3)。
樣本需求量少
目前MeRIP-seq技術(shù)需要≥120g的總RNA起始量,所需樣本量多,限制了MeRIP-seq的適用性。相比于MeRIP-seq,Arraystar表觀轉(zhuǎn)錄組芯片只需1ug 總RNA的起始量,所需RNA的量大大減少(表 1),為珍貴樣本和來源有限的樣本研究RNA修飾提供了機(jī)會(huì)。
| 表觀轉(zhuǎn)錄組芯片 | MeRIP Seq | |
| RNA起始量 | ≥1ug total RNA |
≥120ug total RNA |
| 分離mRNA或去除rRNA | 不需要 |
需要 |
| RNA是否完整 | 不需要 |
需要 |
數(shù)據(jù)庫
Human mRNA&lncRNA Epitranscriptomic microarray
| 探針總數(shù) | 60,613 |
| 探針長度 | 60 nt |
| 探針位點(diǎn) | mRNA & lncRNA: 特異性外顯子和選擇性剪切位點(diǎn) Mid-size ncRNAs: ncRNA的獨(dú)特序列區(qū)域 |
| 探針特異性 | 轉(zhuǎn)錄本特異性 |
| 標(biāo)記方法 | 全長標(biāo)記cRNAs,無3’偏好性,豐度很低的降解RNA也可標(biāo)記 |
| 蛋白編碼mRNAs | 44,122 |
| LncRNAs | 12,496 |
| Mid-size ncRNAs | 1,366 (pre-miRNAs), 1,642 (pri-miRNAs), 19 (snRNAs), 786 (snoRNAs) |
| mRNA 數(shù)據(jù)庫 | Refseq, UCSC, GENECODE, FANTOM5 CAT [2-6] |
| lncRNA 數(shù)據(jù)庫 | Arraystar LncRNA 收集方式: 收錄所有已更新至2018年的數(shù)據(jù)庫和已發(fā)表文獻(xiàn)中的lncRNAs, 每一個(gè)lncRNA基因優(yōu)先挑選“Canonical” 或 “longest” 轉(zhuǎn)錄本 外部數(shù)據(jù)庫 (更新至2018): Refseq, UCSC, GENCODE, FANTOM5 CAT, LncRNAdb, RNAdb, NRED [2-8] 參考文獻(xiàn): 收錄至2018年所有文獻(xiàn)中的lncRNA [9-45] |
| Mid-size ncRNA 數(shù)據(jù)庫 | GENECODE, miRBase [1-2] |
| 芯片規(guī)格 | 8 × 60 K |
Mouse mRNA&lncRNA Epitranscriptomic microarray
| 探針總數(shù) | 60,773 |
| 探針長度 | 60 nt |
| 探針位點(diǎn) | mRNA & lncRNA: 特異性外顯子和選擇性剪切位點(diǎn) Mid-size ncRNAs: ncRNA的獨(dú)特序列區(qū)域 |
| 探針特異性 | 轉(zhuǎn)錄本特異性 |
| 標(biāo)記方法 | 全長標(biāo)記cRNAs,無3’偏好性,豐度很低的降解RNA也可標(biāo)記 |
| 蛋白編碼mRNAs | 48,161 |
| LncRNAs | 8,393 |
| Mid-size ncRNAs | 701 (pre-miRNAs), 957 (pri-miRNAs), 1229 (snRNAs), 1,200 (snoRNAs) |
| mRNA 數(shù)據(jù)庫 | Refseq, UCSC, GENECODE [2-3] |
LncRNA 數(shù)據(jù)庫 |
Arraystar LncRNA 收集方式: 收錄所有已更新至2018年的數(shù)據(jù)庫和已發(fā)表文獻(xiàn)中的lncRNAs, 每一個(gè)lncRNA基因優(yōu)先挑選“Canonical” 或 “longest” 轉(zhuǎn)錄本 外部數(shù)據(jù)庫 (更新至2018): Refseq, UCSC, GENCODE, GenBank, lncRNAdb, NRED [2-7] 參考文獻(xiàn)s: lincRNA分類[8-11], T-UCRs [12], 進(jìn)化保守 LncRNAs [13], 收錄至2018年所有文獻(xiàn)中l(wèi)ncRNA [14-22] |
| Mid-size ncRNA 數(shù)據(jù)庫 | GENECODE, miRBase [1-2] |
| 芯片規(guī)格 | 8 × 60 K |
Rat mRNA&lncRNA Epitranscriptomic microarray
| 探針總數(shù) | 40,991 |
| 探針長度 | 60nt |
| 探針位點(diǎn) | mRNA & lncRNA: 特異性外顯子和選擇性剪切位點(diǎn) Mid-size ncRNAs: ncRNA的獨(dú)特序列區(qū)域 |
| 探針特異性 | 轉(zhuǎn)錄本特異性 |
| 標(biāo)記方法 | 全長標(biāo)記cRNAs,無3’偏好性,豐度很低的降解RNA也可標(biāo)記 |
| 蛋白編碼mRNAs | 27,770 |
| LncRNAs | 10,582 |
| Mid-size ncRNAs | 432 (pre-miRNAs), 449 (pri-miRNAs), 155 (snRNAs), 1,469 (snoRNAs) |
| mRNA 數(shù)據(jù)庫 | Refseq, Ensembl 92[1, 5] |
| LncRNA 數(shù)據(jù)庫 | Arraystar LncRNA 收集方式: 收錄所有已更新至2018年的數(shù)據(jù)庫和已發(fā)表文獻(xiàn)中的lncRNAs, 每一個(gè)lncRNA基因優(yōu)先挑選“Canonical” 或 “longest” 轉(zhuǎn)錄本 正在更新的數(shù)據(jù)庫: Ensembl 92 [1] , Refseq [5] 參考文獻(xiàn): T-UCRs [6-9] |
| Mid-size ncRNA來源數(shù)據(jù)庫 | GENECODE, miRBase, GtRNADb [2-3] |
| 芯片規(guī)格 | 4 × 44K |
實(shí)驗(yàn)流程
Arraystar表觀轉(zhuǎn)錄組芯片提供完整的服務(wù),從樣本準(zhǔn)備,MeRIP,cRNA標(biāo)記,芯片實(shí)驗(yàn)到注釋和數(shù)據(jù)分析。嚴(yán)格的質(zhì)控步驟保證數(shù)據(jù)質(zhì)量和有效性。交給我們您的樣品,我們將做到最好。
m6A-mRNA&lncRNA Epitranscriptomic Microarray實(shí)驗(yàn)流程:
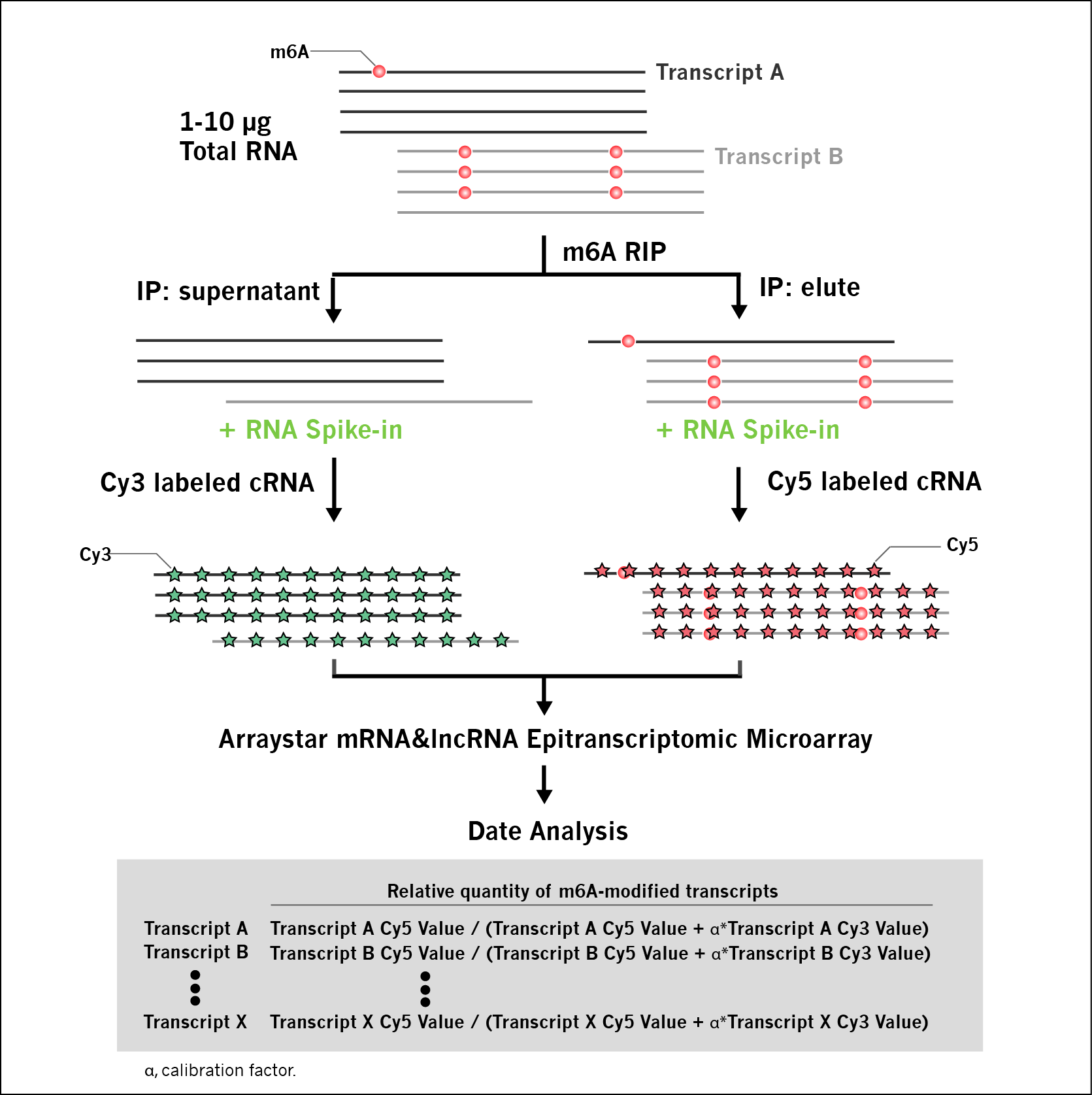
• 客戶提供樣本(具體見樣本指南)
• RNA質(zhì)量檢測
• m6A-RIP
• cRNA合成和雙色熒光標(biāo)記(cy5標(biāo)記IP-RNA,cy3標(biāo)記上清RNA)
• 芯片雜交,洗脫和掃描
• 數(shù)據(jù)采集,數(shù)據(jù)注釋,數(shù)據(jù)分析和總結(jié)
結(jié)果展示
Arraystar在芯片表達(dá)檢測,數(shù)據(jù)分析和結(jié)果注釋方面有著專業(yè)而深入的知識與經(jīng)驗(yàn)。為客戶提供豐富而詳細(xì)的表觀轉(zhuǎn)錄組生物信息學(xué)數(shù)據(jù)分析結(jié)果。
差異m6A甲基化轉(zhuǎn)錄本(mRNA,lncRNA和mid-size ncRNA):
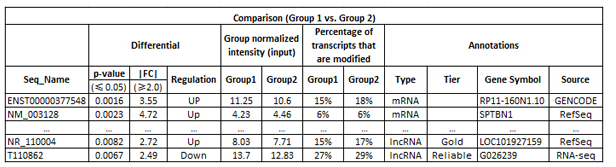
差異m6A甲基化mRNA聚類分析:

差異m6A甲基化mRNA GO富集分析:
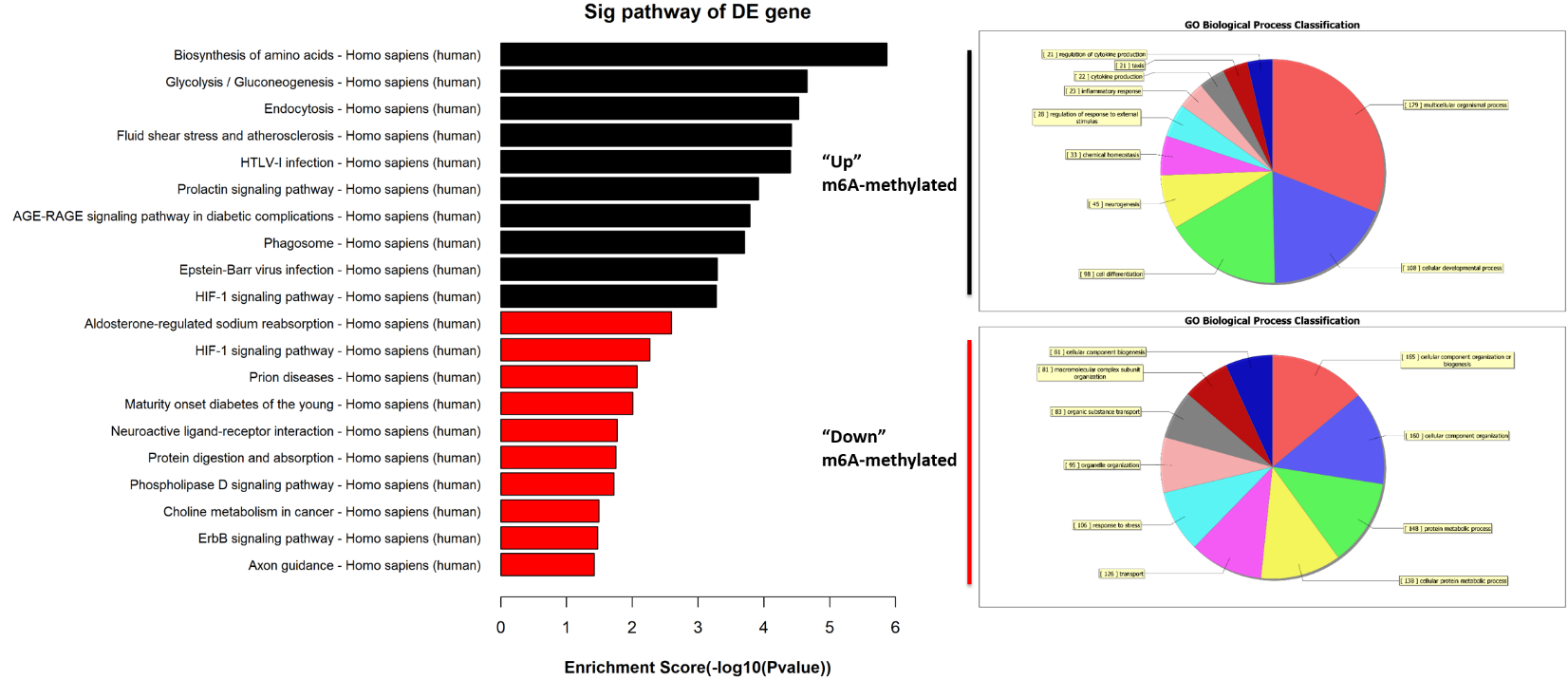
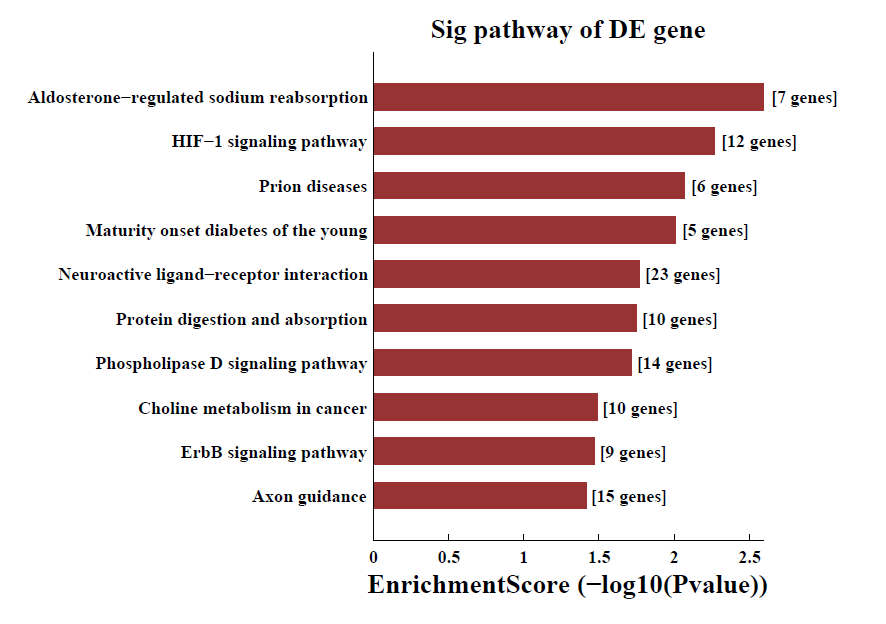
差異m6A甲基化mRNA pathway分析:
- BSI邀您參加蛋白質(zhì)組學(xué)數(shù)據(jù)分析培訓(xùn)會(huì)
- 單細(xì)胞空間多組學(xué)技術(shù)論壇暨廣州站生信培訓(xùn)班報(bào)名中
- SBC單細(xì)胞及空間多組學(xué)實(shí)驗(yàn)與生信分析培訓(xùn)班招生
- 小海龜發(fā)布百個(gè)橫向課題、千篇SCI-生命科學(xué)合作計(jì)劃
- 空間多組學(xué)研究策略及生信分析培訓(xùn)班火熱招生中
- SBC ToolBox云平臺(tái)VIP專區(qū)再添新模塊GSEA數(shù)據(jù)分析
- SBC芯云講壇:國家自然科學(xué)基金的準(zhǔn)備和申報(bào)技巧
- 第十期SBC單細(xì)胞及空間轉(zhuǎn)錄組測序培訓(xùn)班報(bào)名通知
- scMega助力scATAC-seq數(shù)據(jù)與scRNA-seq數(shù)據(jù)的聯(lián)合分析
- 非負(fù)矩陣分解NMF算法助力單細(xì)胞轉(zhuǎn)錄組數(shù)據(jù)分析
- 深度神經(jīng)網(wǎng)絡(luò)助力對腫瘤細(xì)胞鑒定及空間惡性區(qū)域的識別
- 腫瘤純度和倍性評估工具Sequenza的安裝和使用方法
- 單細(xì)胞高級分析百篇文獻(xiàn)結(jié)果展示匯總(六)
- 單細(xì)胞高級分析百篇文獻(xiàn)結(jié)果展示匯總(五)
- 使用SBC ToolBox 單細(xì)胞抖動(dòng)圖模塊美化差異marker基因
- SBC ToolBox在Small RNA-seq定量數(shù)據(jù)分析中的應(yīng)用(下)




