|
利用Chromium 平臺的微流控芯片對微量(1ng)基因組 DNA 進行精確分區(qū),將DNA 片段分配到大量的攜帶特異性 barcode 序列的微滴中,每一個微滴都是一個獨立的反應體系,在微滴中通過 PCR 擴增制備
10× Genomics 文庫,對制備好的文庫在 Illumina 測序平臺上測序。通過將來自同一長片段模板具有相同 barcode 的reads 進行連 接,輸出超長 linked-reads,獲得 DNA 長片段的遺傳信息。
|
||||||||||||||||||
| [發(fā)表評論] [本類其他服務] [本類其他服務商] | ||||||||||||||||||
| 服務商: 北京諾禾致源科技股份有限公司 | 查看該公司所有服務 >> |
Linked-reads技術簡介
10× Genomics 公司的linked-reads 技術本質(zhì)上是利用Chromium 平臺的微流控芯片對微量(1ng)基因組 DNA 進行精確分區(qū),將DNA 片段分配到大量的攜帶特異性 barcode 序列的微滴中,每一個微滴都是一個獨立的反應體系,在微滴中通過 PCR 擴增制備10× Genomics 文庫,對制備好的文庫在 Illumina 測序平臺上測序。通過將來自同一長片段模板具有相同 barcode 的reads 進行連接,輸出超長 linked-reads,獲得 DNA 長片段的遺傳信息。
De novo assembly工作流程
10× Genomics de novo assembly 工作流程包括高分子量DNA 制備、Chromium 平臺進行文庫構建、Illumina 平臺測序以及組裝軟件進行組裝等過程。
1、DNA 樣品制備:總量不低于2μg;主帶大于50Kb,大于100Kb更好。
2、文庫構建:含有 barcode 信息的凝膠珠通過“雙十字”微流體,在第一個連接處結合 DNA 以及酶的混合物,第二個連接處包裹上油滴形成 GEMs 并進行收集。凝膠珠溶解釋放含有 barcode 的序列,隨機引物結合在油滴反應體系內(nèi)長鏈 DNA 模板的隨機位置上進行PCR 擴增,將每個油滴中擴增后獲得的含有 barcode 信息的DNA 片段混合。在片段的另一端連接 Illumina Read 2, 最后通過樣品Index PCR 引入Illumina 測序P5/P7 接頭和樣品index,完成文庫構建。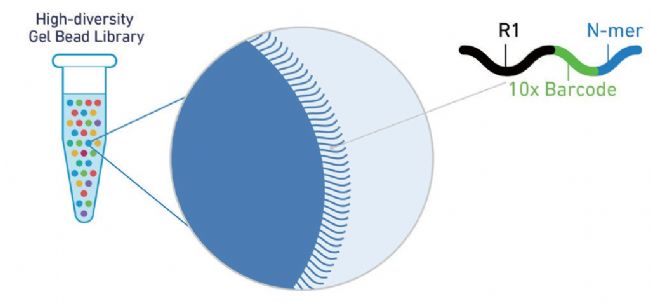
圖1 10× Genomics凝膠珠的特征
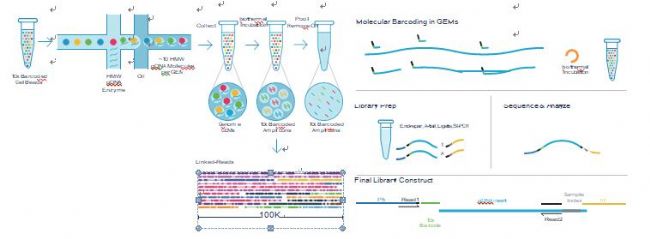
圖2 10× Genomics linked-reads文庫構建
技術優(yōu)勢
10× Genomics de novo assembly 僅需構建一個 10× Genomics 文庫,利用Supernova™ Assembler 軟件即可進行組裝,操作過程簡單,組裝成本低、質(zhì)量高。
1、DNA需求量低:僅需1ng基因組 DNA 即可得到長片段DNA 文庫(原始DNA 量需>2ug)。
2、長跨度信息:根據(jù) barcode 信息將多個Reads 進行拼接,可獲得長達100Kb的跨度信息。
3、組裝周期短:從樣本 DNA 提取到組裝完成僅需2~3周。
4、二倍體組裝:10× Genomics可實現(xiàn)真正的二倍體組裝,揭示樣本特異性序列,發(fā)現(xiàn)真實的基因組信息,研究二倍體基因組結構。
Supernova組裝案例(植物)
應用方向
單個參考基因組的構建
在一個物種已有參考基因組的情況下,對具有重要性狀的栽培種、野Th種開展10× Genomics de novo 測序,可以快速獲得物種高質(zhì)量基因組,挖掘品種特有基因,鑒定重要基因結構變異。
1、辣椒基因組(Horticulture Research,2018)
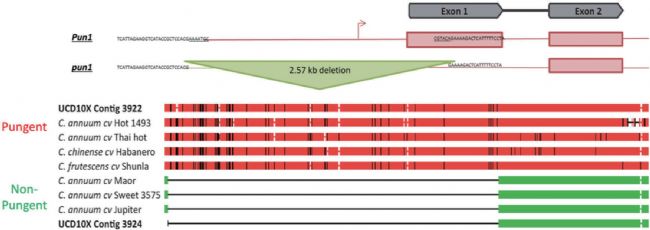
辣椒基因組是一個代表性的復雜植物基因組,基因組大小達3.5Gb,其中75%~80%為重復序列。研究利用10× Genomics linked-reads 技術對一個辣椒雜合 F1 個體進行測序和組裝,最終獲得的基因組大小為3.21Gb,scaffold N50=3.69Mb,組裝得到的序列在準確性和連續(xù)性上優(yōu)于目前所有的辣椒參考基因組。通過 phased 組裝方法,F(xiàn)1 中脂肪酰轉移酶 PUN1 基因中 2.5Kb 的插入/缺失單倍型(代表辣和不辣兩種類型)均被完整地組裝出來。高質(zhì)量的辣椒基因組組裝表明 linked-reads 技術為低成本組裝復雜植物基因組以及通過精確的單倍型構建比較基因結構變異提供了一個途徑。
10× Genomics 公司的linked-reads 技術本質(zhì)上是利用Chromium 平臺的微流控芯片對微量(1ng)基因組 DNA 進行精確分區(qū),將DNA 片段分配到大量的攜帶特異性 barcode 序列的微滴中,每一個微滴都是一個獨立的反應體系,在微滴中通過 PCR 擴增制備10× Genomics 文庫,對制備好的文庫在 Illumina 測序平臺上測序。通過將來自同一長片段模板具有相同 barcode 的reads 進行連接,輸出超長 linked-reads,獲得 DNA 長片段的遺傳信息。
De novo assembly工作流程
10× Genomics de novo assembly 工作流程包括高分子量DNA 制備、Chromium 平臺進行文庫構建、Illumina 平臺測序以及組裝軟件進行組裝等過程。
1、DNA 樣品制備:總量不低于2μg;主帶大于50Kb,大于100Kb更好。
2、文庫構建:含有 barcode 信息的凝膠珠通過“雙十字”微流體,在第一個連接處結合 DNA 以及酶的混合物,第二個連接處包裹上油滴形成 GEMs 并進行收集。凝膠珠溶解釋放含有 barcode 的序列,隨機引物結合在油滴反應體系內(nèi)長鏈 DNA 模板的隨機位置上進行PCR 擴增,將每個油滴中擴增后獲得的含有 barcode 信息的DNA 片段混合。在片段的另一端連接 Illumina Read 2, 最后通過樣品Index PCR 引入Illumina 測序P5/P7 接頭和樣品index,完成文庫構建。

圖1 10× Genomics凝膠珠的特征

圖2 10× Genomics linked-reads文庫構建
3、上機測序:在 Illumina/Hiseq X 平臺上進行雙末端150bp 測序。
4、De novo 組裝:利用10× Genomics 的Supernova™ Assembler 組裝,輸出結果。技術優(yōu)勢
10× Genomics de novo assembly 僅需構建一個 10× Genomics 文庫,利用Supernova™ Assembler 軟件即可進行組裝,操作過程簡單,組裝成本低、質(zhì)量高。
1、DNA需求量低:僅需1ng基因組 DNA 即可得到長片段DNA 文庫(原始DNA 量需>2ug)。
2、長跨度信息:根據(jù) barcode 信息將多個Reads 進行拼接,可獲得長達100Kb的跨度信息。
3、組裝周期短:從樣本 DNA 提取到組裝完成僅需2~3周。
4、二倍體組裝:10× Genomics可實現(xiàn)真正的二倍體組裝,揭示樣本特異性序列,發(fā)現(xiàn)真實的基因組信息,研究二倍體基因組結構。
Supernova組裝案例(植物)
| 物種名稱 | 發(fā)表期刊 | 發(fā)表單位 | 技術策略 | 組裝軟件 | 組裝結果 |
| 玉米B73 Maize B73 |
BMC Genomics 2018.9 |
愛荷華州立大學 |
10×Chromium linked-reads read depth:45× |
supernova Assembler version 1.1.0 |
Contigs Number:234,153 Contig N50=14.5Kb Scaffolds Number: 172,000 Scaffold N50=89Kb Total Size: 1.28Gb Genome Coverage:50% |
| 糜子Panicum miliaceum |
BMC Genomics 2018.9 |
愛荷華州立大學 |
10×Chromium linked-reads read depth:107× |
Supernova Assembler version 1.1.5 |
Scaffolds Number:30,819 Scaffold N50=912Kb Total Size: 823Mb Genome Coverage:83% |
| Contigs Number:134,573 | |||||
| 辣椒Capsicum annuum | Horticulture Research 2018.1 | 加州大學 | 10×Chromium linked-reads read depth:56× | Supernova Assembler version 1.1 | Contig N50= 123Kb Scaffolds Number:83,391 Scaffold N50= 3.69Mb Total Size: 3.21Gb |
| Genome Coverage: 92% | |||||
| Contigs Number:66825 | |||||
| 野Th大豆Glycine latifolia | The Plant Journal 2018.4 | 伊利諾伊大學 | 10×Chromium linked-reads read depth:56× | Supernova Assembler version 1.1.5 | Contig N50=62.61kb Scaffolds Number:42,539 Scaffold N50=853.6 kb Total Size: 939.7Mb Genome Coverage:83.2% |
應用方向
單個參考基因組的構建
在一個物種已有參考基因組的情況下,對具有重要性狀的栽培種、野Th種開展10× Genomics de novo 測序,可以快速獲得物種高質(zhì)量基因組,挖掘品種特有基因,鑒定重要基因結構變異。
1、辣椒基因組(Horticulture Research,2018)
辣椒基因組是一個代表性的復雜植物基因組,基因組大小達3.5Gb,其中75%~80%為重復序列。研究利用10× Genomics linked-reads 技術對一個辣椒雜合 F1 個體進行測序和組裝,最終獲得的基因組大小為3.21Gb,scaffold N50=3.69Mb,組裝得到的序列在準確性和連續(xù)性上優(yōu)于目前所有的辣椒參考基因組。通過 phased 組裝方法,F(xiàn)1 中脂肪酰轉移酶 PUN1 基因中 2.5Kb 的插入/缺失單倍型(代表辣和不辣兩種類型)均被完整地組裝出來。高質(zhì)量的辣椒基因組組裝表明 linked-reads 技術為低成本組裝復雜植物基因組以及通過精確的單倍型構建比較基因結構變異提供了一個途徑。

圖3 F1中PUN1 基因2.5Kb的PAV 被完全重建
2、大豆野Th近緣種基因組(Plant Journal,2018)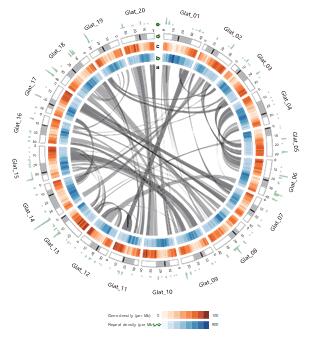
泛基因組研究
與常規(guī)的二代測序組裝方案相比,10× Genomics de novo 只需較少的 DNA 量和較短的組裝周期就可以獲得較好的組裝結果,非常適合于泛基因組研究。
17個人泛基因(Nature Communications,2018)
人類參考基因組被廣泛應用于現(xiàn)代Th物學研究,然而一個參考基因組并不能代表人類群體的完整遺傳信息。研究利用 10× Genomics ed-reads 技術,對來自5個不同人群的17個人(5個非洲人、3個北美人、4個東亞人、3個歐洲人和2個南亞人)進行全基因組測序和 de novo組裝。在17個基因組中鑒定了1842個不存在于參考基因組且唯一的插入變異(non-reference unique insertions,NUIs),總長達2.1Mb。其中64%的NUI 在非人類的靈長類基因組中也有發(fā)現(xiàn),是人類祖先序列;37%的NUI 在人類轉錄組中發(fā)現(xiàn);14% 可能來自于Alu重復序列重組介導的刪除。研究強調(diào)需要一系列包含不同人種的參考基因組來繪制人群完整的遺傳多樣性變異圖譜。
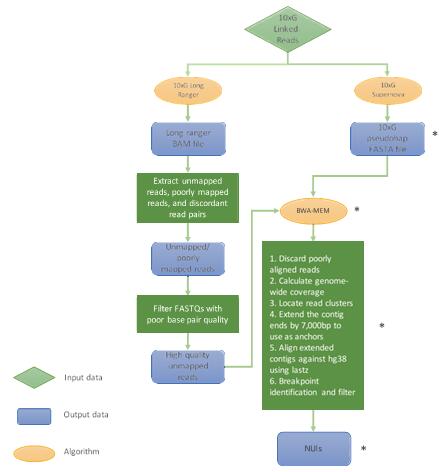
技術策略與組裝指標
參考文獻

Glycine latifolia 是大豆的27 個野Th多年Th 近緣種之一,具有許多大豆不具備的遺傳多樣性和優(yōu)異農(nóng)藝性狀。研究利用10× Genomics linked-reads 技術組裝了939Mb的G. latifolia 基因組草圖。利用遺傳圖譜和大豆基因組序列信息,將 Scaffolds 掛載到了20個染色體級別的假分子中。在組裝基因組中鑒定到了304個NBS-LRR 類抗病蛋白基因和367個參與基礎防御響應和非Th物脅迫響應的 LRR 類受體激酶基因。G. latifolia 基因組的組裝和注釋為促進大豆的遺傳改良提供了寶貴資源。
右圖4 Glycine latifolia 基因組結構
泛基因組研究
與常規(guī)的二代測序組裝方案相比,10× Genomics de novo 只需較少的 DNA 量和較短的組裝周期就可以獲得較好的組裝結果,非常適合于泛基因組研究。
17個人泛基因(Nature Communications,2018)
人類參考基因組被廣泛應用于現(xiàn)代Th物學研究,然而一個參考基因組并不能代表人類群體的完整遺傳信息。研究利用 10× Genomics ed-reads 技術,對來自5個不同人群的17個人(5個非洲人、3個北美人、4個東亞人、3個歐洲人和2個南亞人)進行全基因組測序和 de novo組裝。在17個基因組中鑒定了1842個不存在于參考基因組且唯一的插入變異(non-reference unique insertions,NUIs),總長達2.1Mb。其中64%的NUI 在非人類的靈長類基因組中也有發(fā)現(xiàn),是人類祖先序列;37%的NUI 在人類轉錄組中發(fā)現(xiàn);14% 可能來自于Alu重復序列重組介導的刪除。研究強調(diào)需要一系列包含不同人種的參考基因組來繪制人群完整的遺傳多樣性變異圖譜。

圖5 NUI鑒定的策略和流程
技術策略與組裝指標
| 10× Genomics de novo assembly | |
| 基因組DNA | 總量不低于2μg;主帶大于50Kb,大于100Kb更好 |
| 文庫類型 | 一個10× Genomics文庫 |
| 測序數(shù)據(jù)量 | 120G或180G |
| 組裝指標 | ContigN50>30Kb;ScaffoldN50>1Mb |
參考文獻
- Weisenfeld N I, Kumar V, Shah P, et al. Direct determination of diploid genome sequences[J]. Genome Research, 2017, 27(5):757-767.
- Hulse-Kemp A M , Maheshwari S , Stoffel K , et al. Reference quality assembly of the 3.5-Gb genome of Capsicum annuum from a single linked-read library[J]. Horticulture Research, 2018, 5(1):4.
- Liu Q , Chang S , Hartman G L , et al. Assembly and annotation of a draft genome sequence for Glycine latifolia, a perennial wild relative of soybean[J]. Plant Journal, 2018.
- Wong K H Y , Levysakin M , Kwok P Y . De novo human genome assemblies reveal spectrum of alternative haplotypes in diverse populations[J]. Nature Communications, 2018, 9(1).
Copyright(C) 1998-2025 生物器材網(wǎng) 電話:021-64166852;13621656896 E-mail:info@bio-equip.com


