PacBio單分子測序最新科研動態
“第三代測序技術”的研發已有近十年時間,商業化的第三代測序儀上市也有三年,目前,國內對Pacbio單分子測序研究也有了最新進展:
一,中科院藥植所采用PacBio單分子測序揭示丹參葉綠體DNA修飾之間復雜的相互作用:編碼及非編碼RNA的表達
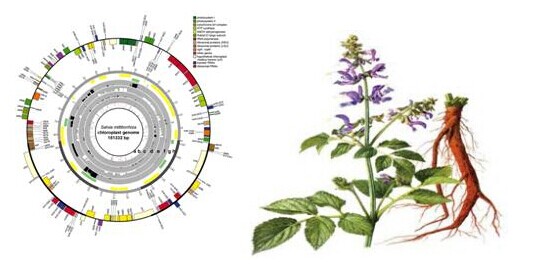
2014年6月10日,中科院藥用植物研究所(IMPLAD)劉昶團隊在《PLOS ONE》雜志上發表了利用
PacBio測序技術揭示丹參(Salvia miltiorrhiza)葉綠體DNA修飾之間復雜相互作用的相關文章,該文章報道了丹參葉綠體中編碼及非編碼RNA的表達情況。這也是國內PacBio第三代測序用戶在國際性雜志發表的第一篇文章。
丹參是最廣泛使用的藥用植物之一。作為基于葉綠體基因工程手段開發使丹參活性成分過表達方法的第一步,該研究團隊從基因組,轉錄組,和堿基修飾三方面對丹參葉綠體進行了分析。先從新鮮葉片中提取總基因組DNA和RNA,然后進行鏈特異性RNA測序和PacBio公司的單分子實時(Single-Molecule Real-Time, SMRT)測序分析。
實驗先是將RNA測序得到的reads mapping到基因組,使該研究小組確定了80個蛋白質編碼基因的相對表達水平。此外,還明確了19個多順反子轉錄單元和136個假定反義和基因間非編碼RNA(ncRNA)基因。將蛋白編碼基因的轉錄本(cRNA)豐度與重疊反義非編碼RNA(asRNA)相比較表明,asRNA的存在與cRNA的豐度增加有關(P<0.05)。使用SMRT Portal軟件預測到了2687個潛在的DNA修飾位點和2個潛在的DNA修飾基序。兩個基序包括TATA盒樣基序(CPGDMM1, ''TATANNNATNA''),以及一個未知的基序(CPGDMM2, ''WNYANTGAW'')。
研究采用二代和三代DNA測序技術并用,使在基因組層面研究非編碼RNA和DNA修飾成為可能。然而,原來關于反義RNA和DNA修飾研究在實驗上具有相當大的困難。首先,大多數asRNA轉錄本表達水平顯著偏低,因而難以用經典技術如Northern Blot和原位雜交進行驗證。第二,正義和反義轉錄本之間錯綜復雜的關系意味著實驗擾動會不可避免地干擾其他轉錄本的表達。因此,通過knocking-in和knocking-out技術確定轉錄本的生物學功能是復雜的。第三,雖然SMRT技術已被證明能夠檢測到潛在的DNA修飾,但驗證這些修飾仍然是個挑戰性的任務。第四,葉綠體asRNA和DNA修飾的存在和功能的驗證是更加困難的。
綜上所述,本研究所描述的一些發現從目前的技術上來講是有巨大進步的。然而,本研究提出的數據已經證實了由asRNA和DNA修飾引起的基因表達調控的復雜性。
二,三代基因測序組裝算法和軟件研發獲突破
“第三代測序技術”的研發已有近十年時間,商業化的第三代測序儀上市也有三年。但目前測序市場仍為二代測序技術所壟斷(我國頂級科研機構和商業公司所擁有的三代測序儀可能僅有數十臺)。三代測序技術產生的讀段更長,測序成本更低,其取代二代技術是測序技術發展的必然趨勢。然而由于三代測序技術錯誤率高,現有的組裝軟件多是對第二代測序數據組裝軟件的“修補”而并沒有充分考慮到三代測序技術的數據特征。事實上,基因組裝算法問題被廣泛認為是計算生物學和生物信息學領域最復雜的計算難題之一,也是目前阻礙基因測序產業從二代技術升級到三代技術最大的技術障礙。
最近,美國馬里蘭大學 Chengxi Ye, James A. Yorke, Aleksey Zimin 等與中國科學院昆明動物研究所遺傳資源與進化國家重點實驗室馬占山研究員在這一領域的合作研發取得新突破。該研究團隊在一篇題為DBG2OLC: Efficient Assembly of Large Genomes Using the Compressed Overlap Graph 的文章中引入了一種新的針對三代測序技術的基因組裝算法,并開發出一款軟件(DBG2OLC)。另外作者(Ye et al. 2011, 2012)于2011年發布的SparseAssembler曾經比當時主流的基因組裝軟件節省90%的內存空間,而其計算時間和組裝質量卻毫不遜色。著名的SOAPdenovo的升級版,也是目前最廣泛應用的基因組裝軟件SOAPdenovo2即采用了SparseAssembler算法。
多組測序數據的測試表明:與目前用于三代測序最優秀的一些基因組裝軟件(例如PacBio2CA, HGAP, ECTools)相比,DBG2OLC在計算時間和內存空間的消耗通常僅為其它算法的1/10。理論上,DBG2OLC 在時間和空間的使用上相對其它同類軟件可減少達1000倍。例如組裝關鍵步驟之一的“兩兩比對”計算,采用一組由 PacBio提供的人類基因組數據,DBG2OLC 使用一臺普通PC僅用了6小時完成。而同樣計算,Pacific Biosciences所報道的時間為 405000 CPU小時,而且是在Google的計算集群上完成。因此,DBG2OLC 算法基本解決了目前三代測序技術所面臨的計算技術挑戰,從而為推進基因測序技術的產業升級奠定了良好的技術基礎。
三,PacBio RS II 測序系統原理
PacBio RS測序儀系統能夠對單個DNA(脫氧核糖核酸)分子進行測序,而目前市場上的主流測序儀只能對分子群體進行平均測序。單分子測序能對DNA中罕見的序列變異進行分析,也不需要在測序之前對DNA樣本進行放大,因為放大過程可能引發錯誤,導致對某個DNA序列檢測失敗。其工作原理是用一種聚合酶將DNA的復制限制在一個微小的間隙中,給各種堿基加上熒光示蹤標記,當堿基合成DNA鏈時,這些熒光標記就會發出不同顏色的閃光,根據閃光顏色就可識別出不同的堿基。
四,PacBio RS II 測序系統特點
1、測序讀長長:平均測序讀長能達到3,000至5,000堿基,最長的序列能達到20,000堿基;
2、準確率高:對基因組組裝和基因組變異檢測,可以最多達到99.999%的準確率;選用特殊測序模式,測序準確率可以在達到單個分子99%準確率的條件下,讀長超過經典的Sanger測序法;
3、極度的敏感性:可以檢測頻率在0.1%的 minor variants;
4、直接檢測廣泛的堿基修飾:除了5-methylcytosine修飾以外, 還可以檢測N6-methyladenine, N4-methylcytosine, DNA氧化損傷 以及其它堿基的修飾.
5、GC偏向性(GC bias)小:在極端高GC和極端低GC區域,可以輕松測定,從而保證序列的均勻覆蓋度;
6、無PCR擴增偏向性:樣本不需要進行PCR擴增,避免了覆蓋度不均一和PCR artifacts.





