naica微滴芯片數字PCR在基因編輯脫靶檢測的應用
導讀
漢堡-埃彭多夫大學醫學中心(UKE)的研究者在Molecular Therapy: Methods & Clinical Development上發表了題為LATE–a novel sensitive cell-based assay for the study of CRISPR/Cas9-related long-term adverse treatment effects的文章。
文中應用流式細胞術和naica®微滴芯片數字PCR系統開發了一種長期不良治療效應(LATE)體外檢測方法評估促生長脫靶事件的風險,通過細胞轉化事件來評估Cas9蛋白的潛在毒性。在小規模原理驗證實驗中,可重復地檢測到由TP53基因脫靶切割引起的原代人新生兒包皮成纖維細胞(NUFF細胞)中低頻的(<0.5%)促生長事件。作者還比較了不同Cas9蛋白的脫靶毒性,證明現有的高保真核酸酶比第一代Cas9脫靶概率大幅降低。在這項概念驗證研究中,LATE檢測可以評估CRISPR/Cas9系統的生理學不良反應,有利于臨床前安全性研究。

自CRISPR / Cas9技術問世以來,基因編輯已廣泛應用于基礎研究和應用生物學領域,將其轉化為臨床試驗則引起了安全性問題。盡管脫靶事件的頻率和位置已被廣泛研究,但人們對它們在大規模、長期的應用環境中潛在的生物學后果知之甚少。所以開發一種操作性強的體外檢測方法來評估這一問題則至關重要。
由于脫靶相關的風險非常明顯,這不僅促成了各種算法的建立來預測靶位點脫靶突變的可能性,而且還建立了幾種無偏、通用的技術來分析全基因組的雙鍵斷裂(DSB)。但現有的主流方法都依賴于某種形式的高通量測序來檢測靶位點或全基因組范圍內的突變。而二代測序(NGS)是一種相當昂貴的方法,也需要先進的生物信息學技術來進行詳細的數據分析。此外,上述所有算法和方法僅能提供脫靶的位置和頻率信息,而它們對細胞生理學的潛在后果仍是未知。目前也缺乏對設計核酸酶Cas9蛋白潛在長期不良影響的評估方法。
應用亮點:
▶ 使用naica®微滴芯片數字PCR系統構建了GEF-dPCR(基因編輯頻率數字PCR)檢測方法。
▶ dPCR在檢測低頻突變時比NGS具有更高靈敏度和準確性。
▶ GEF-dPCR可以和NGS互相驗證,GEF-dPCR可能比NGS更不容易受到單核苷酸缺失或交換的影響。
實驗方法:
之前有研究表明NUFF細胞敲除TP53會導致相對生長優勢,作者根據這種已經確立的生長優勢設計了圖1所示的LATE檢測流程。LATE檢測的步驟如下:
1、通過編碼eGFP、 Cas9和gRNA的一體化慢病毒載體LeGO-CC轉導NUFF細胞;
2、轉導8-10周后每周通過流式細胞術分析細胞的熒光強度;
3、通過定量轉導細胞生長優勢指標來判斷基因組編輯的影響,陽性結果中熒光細胞占絕對優勢,陰性結果中熒光細胞不存在生長優勢。
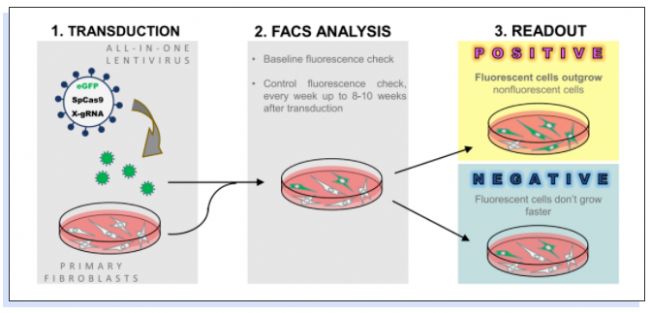
▲圖1. LATE檢測原理圖
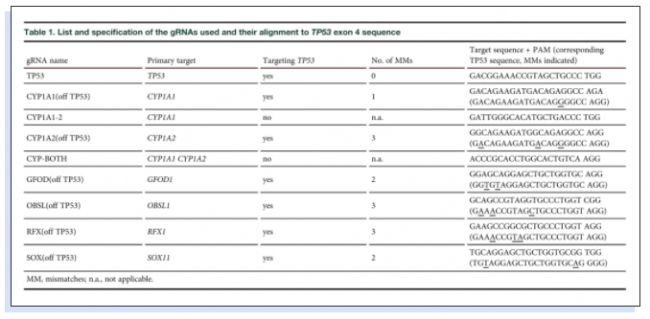
為了確定LATE檢測是否也可用于評估由非預期脫靶TP53敲除引起的生長優勢,作者篩選了5個靶向其他基因但與TP53具有一定相似性的gRNA,它們含有1到3個與TP53編碼序列的錯配。如表1所示,CYP1A1/2、GFOD、OBSL和RFX gRNA會有一定概率靶向TP53,引起NUFF細胞的生長優勢。
表1.研究使用的gRNA列表及其與TP53外顯子4序列的比對
LATE檢測中的陽性結果理論上也可能是由非預期脫靶TP53敲除以外的原因(如插入突變)引起的。為了研究LATE檢測的陽性結果和(從頭產生的)TP53插入/缺失(indel)之間的聯系,作者使用naica®微滴芯片數字PCR系統設計了一種新型的GEF-dPCR技術,又被稱為drop-off檢測。該方法使用兩個雙標記的水解探針,可用于定量Cas9介導的DSB修復期間由非同源末端連接(NHEJ)引起的gRNA結合位點的indel頻率。HEX標記的探針結合區域遠離gRNA識別序列,因此其結合不受DNA修復過程影響。FAM標記drop-off探針的結合會被NHEJ產生的indel影響。

實驗結果:
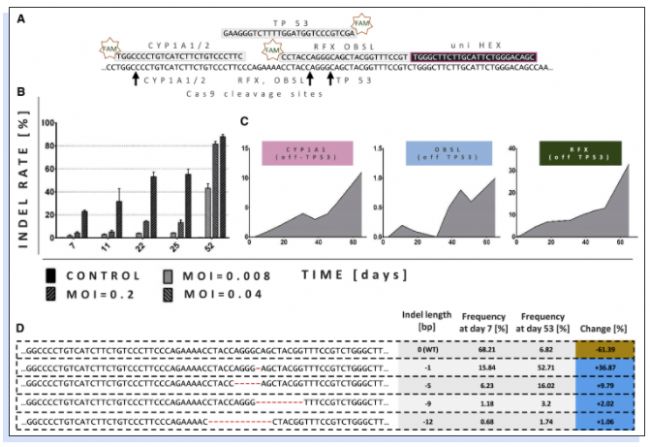
作者應用GEF-dPCR來量化編碼Cas9和CYP1A1、OBSL和RFX gRNA的LeGO CC載體轉導NUFF細胞中脫靶至TP53引入的indel頻率。他們使用了相同的PCR引物和HEX標記的探針,并設計了FAM標記的靶標特異性drop-off探針(圖3A)。結果顯示三種gRNA的陽性LATE檢測結果確實與TP53 indel的增加相關(圖3C)。值得注意的是,在轉導OBSL gRNA的情況下,可檢測到的TP53 indel頻率低至0.2%(圖3C),這種低頻的脫靶效應可以通過LATE檢測來定量,并且在所有對照樣本中沒有檢測到類似的TP53外顯子4 突變和indel。這一觀察凸顯了LATE檢測在發現罕見的促生長事件方面的敏感性,比NGS具有更高靈敏度和準確性。
點擊圖片查看大圖
▲圖3 NUFF細胞獲得的生長優勢與TP53中的indel頻率相關。(A)用于GEF-dPCR的TP53外顯子4和互補的FAM和HEX標記探針片段如圖所示。FAM標記的探針結合gRNA靶向Cas9的位置,因此當TP53序列的互補區域內沒有indel時能夠結合。HEX標記探針結合Cas9切割位點外的TP53序列。(B)在指定時間點從NUFF細胞中獲得基因組DNA樣品,這些細胞在三個不同的MOI下轉導了編碼eGFP,Cas9和靶向TP53 gRNA(LeGO-CC-p53)的一體化慢病毒顆粒(LeGO-CC-p53)。通過GEF-dPCR定量一段時間內靶向TP53外顯子4的indel頻率。(C)在指定的時間點從NUFF細胞中獲得基因組DNA樣品,這些細胞轉導了編碼靶向Cas9及CYP1A1,OBSL1或RFX1的gRNA一體化慢病毒顆粒。通過GEF-dPCR定量一段時間內非預期脫靶TP53外顯子4敲除的indel頻率。(D)用LeGO-CC-p53治療后第7天和第53天對TP53外顯子4進行深度測序得到的indel長度及頻率。
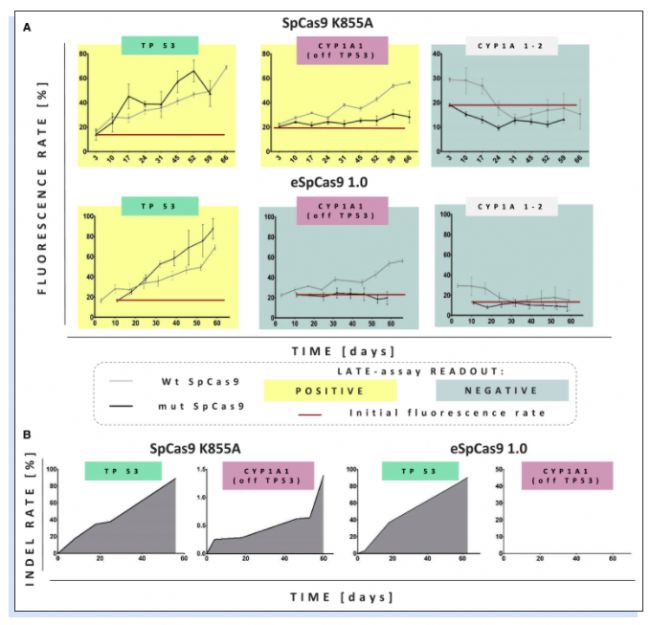
為了增加CRISPR/Cas9系統的靶向特異性,Cas9核酸酶一直是優化的重點。科學家對SpCas9一直在進行結構設計的修改以及隨機誘變。有研究表明正電荷殘基(K855A)的單個丙氨酸取代可以降低Cas9脫靶活性。作者通過病毒轉導靶向TP53分析SpCas9 K855A的核酸酶特異性。如圖6A所示,在實驗前10天,經SpCas9 K855A和wt Cas9處理細胞的eGFP陽性細胞生長動力學接近,但隨著時間的推移SpCas9 K855A處理的eGFP陽性細胞數量更多。作者通過GEF-dPCR和深度擴增子測序這兩種技術都證實,隨著時間的推移TP53中攜帶indel的細胞數量不斷增加。GEF-dPCR檢測到轉導52天后最高插入indel頻率為65%(圖6B),在轉導后第55天約80%的NGS讀數檢測到indel。有趣的是當SpCas9 K855A與CYP1A1(非靶向TP53)gRNA一起使用時,與用相同gRNA和wt Cas9處理的NUFF細胞相比,eGFP陽性細胞的比例生長慢得多(圖6A),這應該與SpCas 9 K855A特異性的提高有關。
在第二組實驗中,作者對eSpCas9 1.0(特異性優于SpCas 9 K855A)進行了上述相同的分析,在LATE試驗中沒有觀察到CYP1A1 gRNA脫靶TP53的eGFP陽性細胞的生長,而靶向TP53的gRNA與wt Cas9一樣有效(圖6A)。
點擊圖片查看大圖
▲圖6 LATE檢測區分具有不同保真度的 SpCas9變體。(A)NUFF細胞與編碼野生型wt SpCas9(灰色)或存在突變位點的SpCas9(黑色)的多合一LeGO-CC顆粒轉導的NUFF細胞的FC分析比較。紅線標記編碼突變 SpCas9 變體的載體的初始轉導率。陽性和陰性分別用黃色和藍灰色表示。(B) 通過 GEF-dPCR 檢測靶向TP53和非靶向 TP53(CYP1A1) 外顯子 4的indel隨時間推移的相對數量。
結論:
這里介紹的LATE檢測結合GEF-dPCR技術可以填補基因編輯中的部分空白,作為一種簡單,快速和高性價比的方法,用于測試給定基因組編輯策略對細胞生長調節的不良影響,評估不同的Cas9核酸酶在基因組編輯時的特異性。
原文鏈接如下:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8399046/#bib33
期刊介紹:
《molecular therapy》是發布分子和細胞療法在糾正遺傳和獲得性疾病研究成果的國際領先期刊。文章涉及基因轉移和編輯、載體開發和設計、干細胞操作、疫苗開發、臨床前靶點驗證、安全性/有效性研究和臨床試驗方面等研究領域。





