WGBS等在揭示植物基因體動態DNA甲基化與基因表達可塑性相關的應用
在一些真核生物中,DNA甲基化發生在基因編碼區,稱為基因體甲基化(gene body methylation,GbM)。盡管DNA甲基化在轉座子和重復DNA沉默中的作用已得到很好的表征,但基因體甲基化與轉錄抑制無關,其生物學重要性尚不清楚。
2023年10月12日,美國加州大學伯克利分校植物與微生物生物學系Ben P. Williams團隊在《Genome Biology》雜志上發表題為“Dynamic DNA methylation turnover in gene bodies is associated with enhanced gene expression plasticity in plants”的研究論文,該研究以模式植物擬南芥為研究對象,通過WGBS、EM-seq、RNA-seq等多組學分析揭示了植物genebody中DNA甲基化動態變化與基因表達可塑性增強相關。

標題:Dynamic DNA methylation turnover in gene bodies is associated with enhanced gene expression plasticity in plants(植物genebody中DNA甲基化動態變化與基因表達可塑性增強相關)
時間:2023-10-12
期刊:Genome Biology
影響因子:12.3 / 1區
技術平臺:WGBS、EM-seq、RNA-seq、數據庫ChIP-seq分析等
研究摘要:
本研究揭示了一種在植物中新發現的基因體甲基化(GbM)類型,它在包括種系在內的所有細胞中均受動態甲基化修飾的組成性添加和去除。動態GbM基因甲基化通過植物特異性DNA去甲基化酶家族(統稱為DRDD酶)去甲基化通路去除,并通過未知的de novo甲基化來源添加(最可能是維持甲基轉移酶MET1)。分析結果揭示動態GbM狀態存在于跨越1億多年的不同譜系同源基因中,表明了進化保守性。與其他基因體甲基化基因相比,動態GbM與基因體內啟動子或調節染色質狀態的存在密切相關。動態GbM與發育和不同生理條件下基因表達可塑性增強有關,而穩定甲基化的GbM基因表現出可塑性降低。動態GbM基因在drdd突變體中表現出降低的動態變化,表明DNA去甲基化和基因表達可塑性增強之間存在因果關系。
本研究提出了一種新的GbM調節基因表達可塑性模型(包括一種新型的GbM),其中基因表達塑性的增加與DNA甲基化writers和erasers活性以及調節染色質狀態富集相關。
材料方法:
材料:
擬南芥:WT野生型vs DRDD突變體植物
研究結果:
(1)DNA去甲基化靶向基因體子集
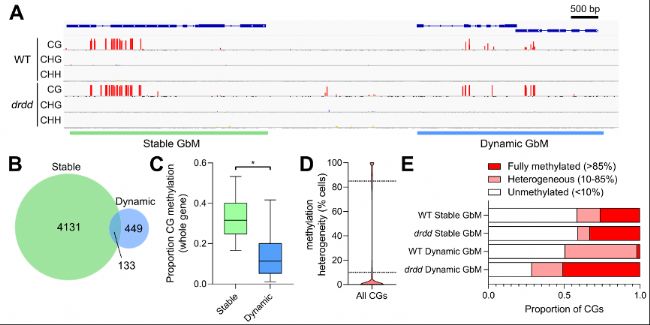 圖1:擬南芥基因組中兩種不同類型的基因體甲基化(GbM)。
圖1:擬南芥基因組中兩種不同類型的基因體甲基化(GbM)。
(2)DNA去甲基化在所有組織和細胞類型中表現出細胞異質性
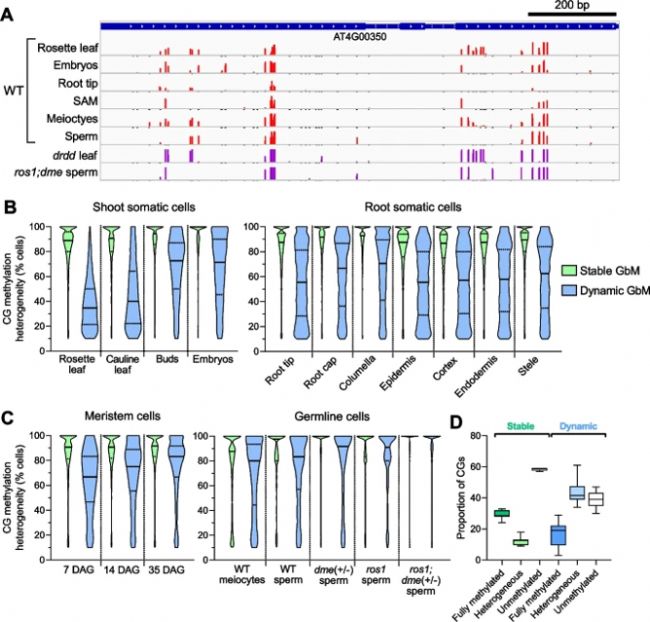 圖2:動態GbM基因在所有細胞和組織類型中都表現出甲基化異質性。
圖2:動態GbM基因在所有細胞和組織類型中都表現出甲基化異質性。
D. 穩態和動態GbM基因體中所有組織和細胞類型中完全甲基化、異質性甲基化和未甲基化CGs比例
(3)需要De novo甲基化來維持動態GbM
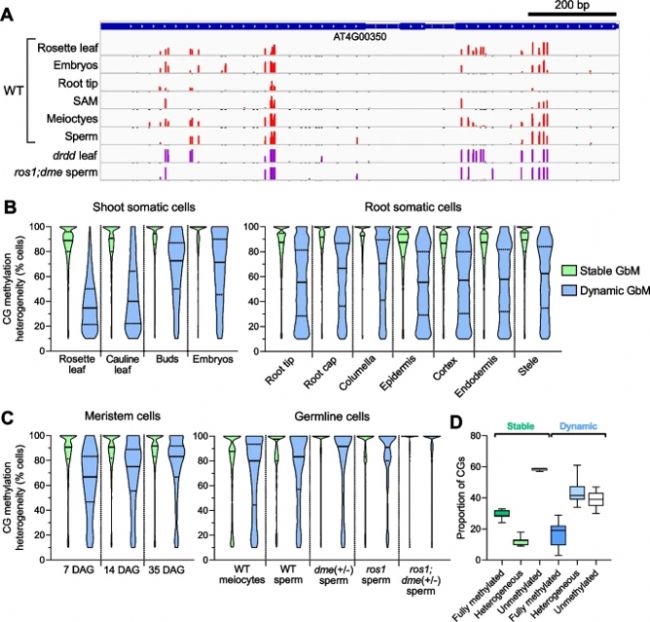
圖2:動態GbM基因在所有細胞和組織類型中都表現出甲基化異質性。
D. 穩態和動態GbM基因體中所有組織和細胞類型中完全甲基化、異質性甲基化和未甲基化CGs比例
(3)需要De novo甲基化來維持動態GbM
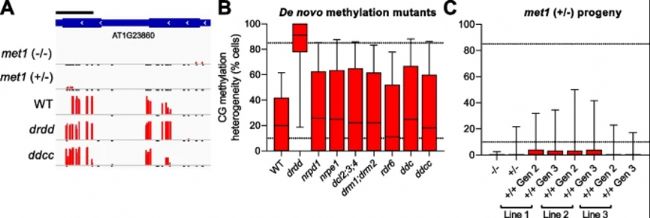 圖3:動態GbM狀態由MET1維持。
圖3:動態GbM狀態由MET1維持。
(4)動態GbM位點在遠緣譜系中保守
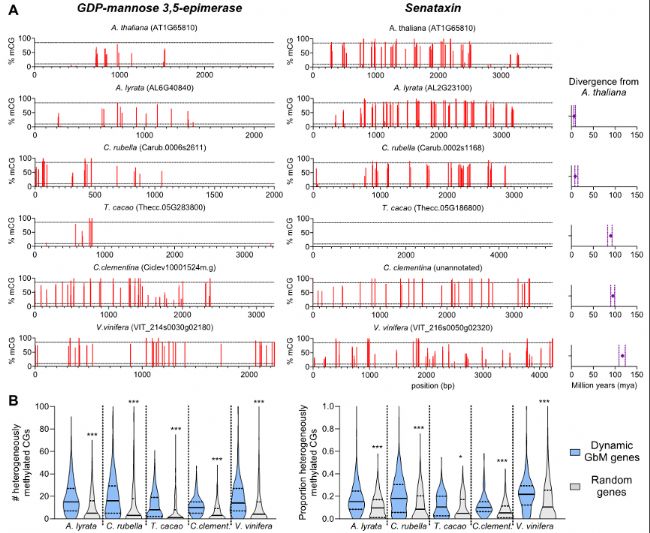
圖4:動態GbM狀態的進化保守。
(5)動態GbM與調節染色質狀態和增強基因表達可塑性有關
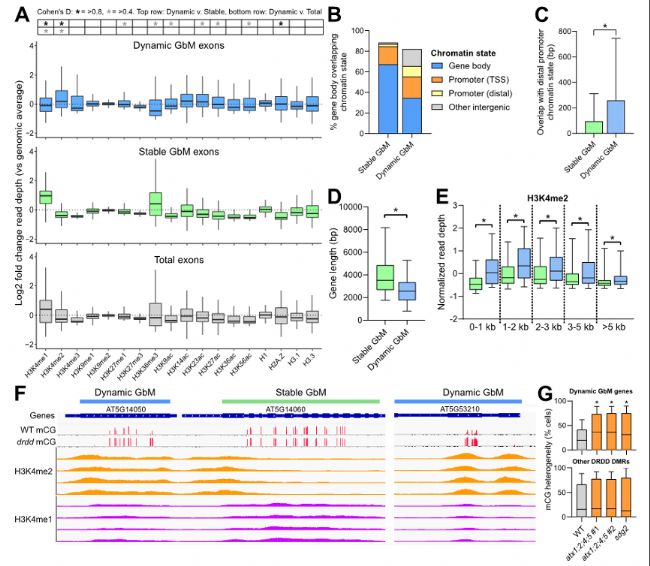 圖5:動態GbM與調節染色質標記之間的相關性。
圖5:動態GbM與調節染色質標記之間的相關性。
(6)動態GbM與基因表達可塑性增加有關
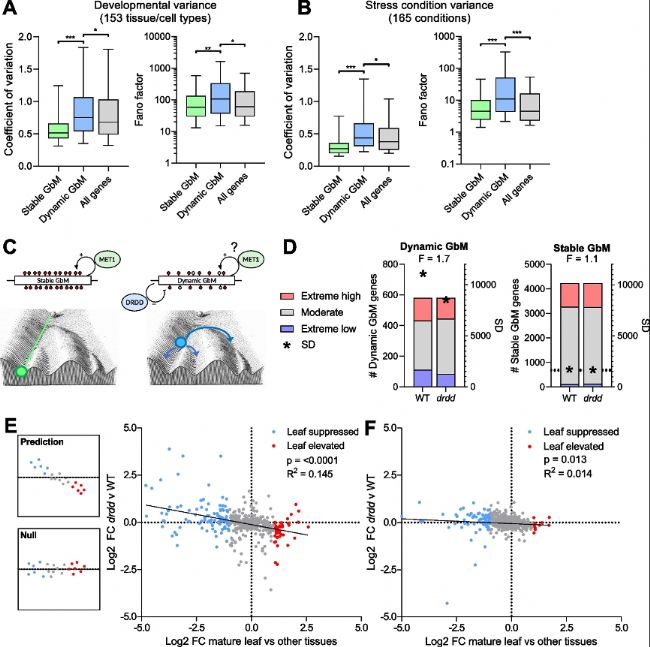 圖6:動態GbM與基因表達可塑性增加有關。
圖6:動態GbM與基因表達可塑性增加有關。
A-B. 箱型圖顯示了所有基因、穩態GbM和動態GbM基因在發育(A)和不同生理應激條件(B)下的變異系數和Fano因子表達水平。
C. 雙尾t檢驗顯示穩態和動態GbM對基因表達可塑性的影響示意圖,以Waddington's landscape表示。穩態GbM基因表現出低方差,表明其單一、穩健的基因表達狀態。動態GbM基因表現出高方差,表明其可能呈現多種可能的基因表達狀態。
D. 動態和穩定的GbM基因在WT和drdd突變體中的高表達和低表達十分位數中的比例。動態和穩定基因的標準差用星號表示。F表示方差相等的F檢驗(動態GbM p=<0.0001,穩態GbM p=0.1)。
E. 散點圖顯示與WT和drdd葉片之間的log2倍數變化相比,WT中葉片與其他組織之間的動態GbM基因表達的log2倍數變化(x軸)。基因表達可塑性喪失被預測為具有顯著的負相關。
F. 比較穩態GbM基因對照組的等效倍數變化值散點圖。R2、p值和線性回歸模型的最佳擬合線
研究結論
本研究定義了植物表觀遺傳學的新表征。基因組中的子集基因體是所有細胞中DNA甲基化writers和erasers標靶,從而產生動態的DNA甲基化變化。這種動態的DNA甲基化狀態與“啟動子樣”染色質狀態以及增強的基因表達可塑性相關。此外,研究揭示了典型的基因體甲基化(在所有細胞中都呈穩態)和低可塑性的通道化表達狀態之間的明確聯系。總的來說本研究為理解真核生物中神秘基因體甲基化的功能作用提供了實質性的進展。
參考文獻:
Williams CJ, Dai D, Tran KA, Monroe JG, Williams BP. Dynamic DNA methylation turnover in gene bodies is associated with enhanced gene expression plasticity in plants. Genome Biol. 2023 Oct 12;24(1):227.
2023年10月12日,美國加州大學伯克利分校植物與微生物生物學系Ben P. Williams團隊在《Genome Biology》雜志上發表題為“Dynamic DNA methylation turnover in gene bodies is associated with enhanced gene expression plasticity in plants”的研究論文,該研究以模式植物擬南芥為研究對象,通過WGBS、EM-seq、RNA-seq等多組學分析揭示了植物genebody中DNA甲基化動態變化與基因表達可塑性增強相關。

標題:Dynamic DNA methylation turnover in gene bodies is associated with enhanced gene expression plasticity in plants(植物genebody中DNA甲基化動態變化與基因表達可塑性增強相關)
時間:2023-10-12
期刊:Genome Biology
影響因子:12.3 / 1區
技術平臺:WGBS、EM-seq、RNA-seq、數據庫ChIP-seq分析等
研究摘要:
本研究揭示了一種在植物中新發現的基因體甲基化(GbM)類型,它在包括種系在內的所有細胞中均受動態甲基化修飾的組成性添加和去除。動態GbM基因甲基化通過植物特異性DNA去甲基化酶家族(統稱為DRDD酶)去甲基化通路去除,并通過未知的de novo甲基化來源添加(最可能是維持甲基轉移酶MET1)。分析結果揭示動態GbM狀態存在于跨越1億多年的不同譜系同源基因中,表明了進化保守性。與其他基因體甲基化基因相比,動態GbM與基因體內啟動子或調節染色質狀態的存在密切相關。動態GbM與發育和不同生理條件下基因表達可塑性增強有關,而穩定甲基化的GbM基因表現出可塑性降低。動態GbM基因在drdd突變體中表現出降低的動態變化,表明DNA去甲基化和基因表達可塑性增強之間存在因果關系。
本研究提出了一種新的GbM調節基因表達可塑性模型(包括一種新型的GbM),其中基因表達塑性的增加與DNA甲基化writers和erasers活性以及調節染色質狀態富集相關。
材料方法:
材料:
擬南芥:WT野生型vs DRDD突變體植物

研究結果:
(1)DNA去甲基化靶向基因體子集
 圖1:擬南芥基因組中兩種不同類型的基因體甲基化(GbM)。
圖1:擬南芥基因組中兩種不同類型的基因體甲基化(GbM)。
- 基因組瀏覽器圖顯示WT和drdd突變體中穩態GbM基因和動態GbM基因的代表性示例。紅色條形圖表示甲基化胞嘧啶,條形圖高度表示胞嘧啶甲基化細胞百分比(比例=0–100%)。
- 穩態和動態GbM基因的數量以及兩組之間的重疊維恩圖。
- 穩態或動態GbM的整體基因(所有細胞的所有CG)中CG甲基化的總體百分比箱形圖。線=中位數;框=四分位數范圍;whiskers=第5個百分位和第95個百分位;*p=<0.0001(雙尾t檢驗)。
- 擬南芥基因組中所有CG的甲基化異質性水平分布小提琴圖,顯示為CG甲基化的細胞百分比。
- 在WT和drdd突變體中,穩態和動態GbM基因體內完全甲基化、異質性甲基化和未甲基化CG比例
(2)DNA去甲基化在所有組織和細胞類型中表現出細胞異質性
 圖2:動態GbM基因在所有細胞和組織類型中都表現出甲基化異質性。
圖2:動態GbM基因在所有細胞和組織類型中都表現出甲基化異質性。
- 代表性的基因組瀏覽器快照顯示在各種細胞和組織類型的動態GbM位點的異質甲基化CGs(比例=0–100%)。
D. 穩態和動態GbM基因體中所有組織和細胞類型中完全甲基化、異質性甲基化和未甲基化CGs比例
(3)需要De novo甲基化來維持動態GbM

圖2:動態GbM基因在所有細胞和組織類型中都表現出甲基化異質性。
- 代表性的基因組瀏覽器快照顯示在各種細胞和組織類型的動態GbM位點的異質甲基化CGs(比例=0–100%)。
D. 穩態和動態GbM基因體中所有組織和細胞類型中完全甲基化、異質性甲基化和未甲基化CGs比例
(3)需要De novo甲基化來維持動態GbM
 圖3:動態GbM狀態由MET1維持。
圖3:動態GbM狀態由MET1維持。
- met1、ddcc和drdd突變體中動態GbM位點的代表性基因組瀏覽器快照。比例尺=200 bp。
- 箱形圖顯示WT、drdd突變體和多個de novo甲基化突變體中動態GbM基因內CGs細胞異質性。
- 顯示純合(homozygous)(−/−) 和雜合(heterozygous )(+/−) met1突變體以及自身met1雜合子的多個WT分段子代。在B和C中,水平線=中值;框=四分位范圍;須(whiskers)=第95個百分位。水平虛線表示用于鑒定非均勻甲基化CGs的臨界值(所有細胞中甲基化率為10-85%)
(4)動態GbM位點在遠緣譜系中保守

圖4:動態GbM狀態的進化保守。
- 六個不同物種的具有代表性的動態GbM基因同源物。每個紅條代表單CG,條高是細胞間甲基化異質性百分比。虛線代表calling異質性甲基化胞嘧啶(10-85%的細胞)臨界值。最右邊panel:物種和擬南芥之間的估計分化時間,來自TimeTree。點表示中值發散時間估計,垂直虛線表示95%置信區間。
- 小提琴圖顯示了動態GbM基因的所有同源物和隨機選擇的十個相同大小的基因組中異質甲基化CGs的數量(左圖)和比例(右圖)。實線表示中位數,虛線表示四分位范圍;*p=<0.05,***p=<0.0005(雙尾t檢驗)
(5)動態GbM與調節染色質狀態和增強基因表達可塑性有關
 圖5:動態GbM與調節染色質標記之間的相關性。
圖5:動態GbM與調節染色質標記之間的相關性。
- 箱形圖顯示了與動態GbM結構域(藍色)、穩態GbM結構區(綠色)或總外顯子(灰色)重疊的外顯子內14個組蛋白修飾的ChIP-seq數據的歸一化reads深度。
- 穩態和動態GbM基因體與鑒定的四種染色質狀態之間的重疊百分比。
- 在穩態和動態GbM基因體之間重疊的總堿基對,以及在遠端啟動子區富集的染色質狀態。
- 顯示穩態和動態GbM基因的基因體長度分布箱形圖。
- 箱形圖顯示基于基因體長分離到bins中穩態和動態GbM基因的H3K4me2-ChIP-seq數據的歸一化reads深度。C、D、E、*p=<0.0001(雙尾參數t檢驗)。
- 穩態和動態GbM基因的H3K4me1和H3K4me2 ChIP-seq原始數據的基因組瀏覽器快照。軌跡代表四項獨立研究的WT數據。
- 箱型圖顯示WT和兩種H3K4me2修飾atx1/atx2/atx4/atx5和sdg2降低的突變背景中動態GbM基因甲基化結構域內CGs的細胞異質性; *p=<0.0001(非參數雙尾t檢驗)
(6)動態GbM與基因表達可塑性增加有關
 圖6:動態GbM與基因表達可塑性增加有關。
圖6:動態GbM與基因表達可塑性增加有關。A-B. 箱型圖顯示了所有基因、穩態GbM和動態GbM基因在發育(A)和不同生理應激條件(B)下的變異系數和Fano因子表達水平。
C. 雙尾t檢驗顯示穩態和動態GbM對基因表達可塑性的影響示意圖,以Waddington's landscape表示。穩態GbM基因表現出低方差,表明其單一、穩健的基因表達狀態。動態GbM基因表現出高方差,表明其可能呈現多種可能的基因表達狀態。
D. 動態和穩定的GbM基因在WT和drdd突變體中的高表達和低表達十分位數中的比例。動態和穩定基因的標準差用星號表示。F表示方差相等的F檢驗(動態GbM p=<0.0001,穩態GbM p=0.1)。
E. 散點圖顯示與WT和drdd葉片之間的log2倍數變化相比,WT中葉片與其他組織之間的動態GbM基因表達的log2倍數變化(x軸)。基因表達可塑性喪失被預測為具有顯著的負相關。
F. 比較穩態GbM基因對照組的等效倍數變化值散點圖。R2、p值和線性回歸模型的最佳擬合線
研究結論
本研究定義了植物表觀遺傳學的新表征。基因組中的子集基因體是所有細胞中DNA甲基化writers和erasers標靶,從而產生動態的DNA甲基化變化。這種動態的DNA甲基化狀態與“啟動子樣”染色質狀態以及增強的基因表達可塑性相關。此外,研究揭示了典型的基因體甲基化(在所有細胞中都呈穩態)和低可塑性的通道化表達狀態之間的明確聯系。總的來說本研究為理解真核生物中神秘基因體甲基化的功能作用提供了實質性的進展。
參考文獻:
Williams CJ, Dai D, Tran KA, Monroe JG, Williams BP. Dynamic DNA methylation turnover in gene bodies is associated with enhanced gene expression plasticity in plants. Genome Biol. 2023 Oct 12;24(1):227.
標簽:
WGBS
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com





