ChIP-seq等在揭示植物H3K27me3相關轉座元件沉默模式中的應用
轉座元件(transposable elements,TEs)是基因組中能夠移動和復制的重復序列,其活動通常對宿主生物有害,因此被宿主嚴格調控。然而,TEs的活動在進化中也扮演了重要角色,例如通過為附近基因提供遺傳或表觀遺傳調控模塊來影響轉錄程序。TEs沉默通常與高DNA甲基化相關,而在植物中,TEs的甲基化可以發生在CG、CHG和CHH三種序列環境中,并與組蛋白H3K9二甲基化(H3K9me2)耦合,這兩種表觀遺傳標記共同抑制TEs的表達和轉座,從而保證基因組完整性。另一方面,H3K27me3與基因轉錄抑制相關,通常與發育、生殖或代謝和應激反應相關的蛋白編碼基因有關。盡管H3K27me3和DNA甲基化通常被認為是互斥的系統,分別特異性靶向基因和TEs的轉錄沉默,但有研究表明在某些情況下,這兩種標記可以共存并協同作用。
近日,法國巴黎薩克雷大學細胞綜合生物學研究所Angélique Déléris團隊以模式生物擬南芥為研究對象,研究了植物中轉座元件(TEs)的表觀遺傳沉默機制,特別是與H3K27me3(組蛋白H3K27三甲基化)相關的沉默狀態。研究結果揭示了在野生型擬南芥中,許多TEs可以被H3K27me3單獨靶向并沉默(而不依賴DNA甲基化沉默機制)。此外,研究還發現了TEs在不同擬南芥自然生態型之間存在表觀遺傳狀態的轉換,即從DNA甲基化到H3K27me3的相互轉換,這一現象被稱為“bifrons”。相關研究成果以“Alternative silencing states of transposable elements in Arabidopsis associated with H3K27me3”為題發表于《Genome Biology》(IF:10.1)期刊。

標題:Alternative silencing states of transposable elements in Arabidopsis associated with H3K27me3(擬南芥中與 H3K27me3 相關的轉座因子可變沉默狀態)
發表時間:2025-01-20
期刊:Genome Biology
影響因子:IF10.1/Q1
技術平臺:ChIP-seq、BS
研究摘要
DNA/H3K9甲基化和Polycomb蛋白組(Polycomb-group,PcG)-H3K27me3沉默通路長期以來被認為在哺乳動物、植物和真菌中是互斥的,并且分別特異性地作用于轉座元件(TEs)和基因。但即使在DNA/H3K9甲基化機制缺失情況下,許多TEs仍然可以被H3K27me3靶向,有時甚至與DNA甲基化共存。
本研究結果揭示了在野生型擬南芥植物中,TEs也可以被H3K27me3單獨靶向并沉默。這些被H3K27me3標記的TEs不僅包括退化殘跡,還包括看似完整的拷貝,它們顯示出響應性PcG靶基因的表觀遺傳特征以及活躍的H3K27me3調控。同時,H3K27me3可以以TE特異性方式沉積在新插入的轉基因TE序列上,表明沉默由順式( cis)作用決定。最后通過比較擬南芥的自然生態型發現了一類TEs(稱為“bifrons”),它們的標記取決于生態型,要么被DNA甲基化標記,要么被H3K27me3標記。這種差異可以追溯到TEs的內在特征以及反式作用因子,并揭示TEs在其生命周期中表觀遺傳狀態變化。
總之,本研究揭示了開花植物中與H3K27me3相關的可變TE沉默模式,同時還表明了在物種水平上兩種表觀遺傳標記(DNA甲基化和H3K27me3)之間存在動態轉換,這一新的范式可能擴展到其他多細胞真核生物。
研究方法
(1)ChIP-seq技術:檢測H3K27me3在TEs上的分布情況。
(2)BS技術:用于分析TEs的DNA甲基化水平。
結果圖形
(1)在野生型擬南芥植物中,許多TE只由H3K27me3標記
研究發現,擬南芥基因組中有超過4000個TEs被H3K27me3標記,這些TEs不僅包括退化的殘跡,還包括看似完整的拷貝。這些TEs顯示出與響應環境或發育信號的經典PcG靶基因相似的染色質特征,其H3K27me3模式受到組蛋白H3K27去甲基化酶的積極調控。
根據H3K27me3和DNA甲基化的覆蓋長度,將TEs分為四類。其中,“class 3”僅被H3K27me3標記,占TE總數的15%,但僅占TE堿基的8.4%。這些TEs傾向于分布在短TE類別中(尤其是小于1kb的TE),而長TEs(大于3.5kb)通常與DNA甲基化相關,盡管一小部分長TEs(大于3.5kb)僅被H3K27me3標記。
這些TEs在基因組中的分布與DNA甲基化的TEs不同,它們不僅存在于異染色質區域,還分散在常染色質臂和近著絲粒區域。
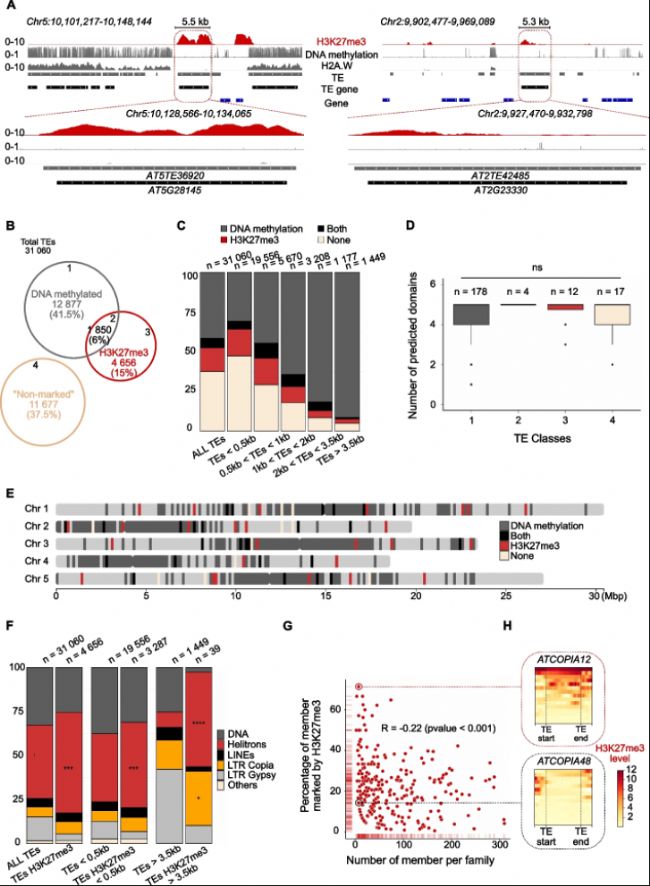
圖1:在野生型擬南芥(Col-0)中,許多轉座元件(TEs)被H3K27me3標記。
A. 代表性基因組瀏覽器視圖顯示野生型Col-0中兩個被H3K27me3標記的TEs的H3K27me3(紅色)、DNA甲基化(灰色)和組成型異染色質組蛋白變體H2A.W(灰色)水平。
B. 基于表觀遺傳修飾的TE分類的維恩圖。當CG甲基化在TE長度上的平均值>50%時,TE被定義為“DNA甲基化”(類別1-2)。當H3K27me3 peaks至少覆蓋TE長度的20%時,TE被定義為處于H3K27me3狀態。
C. 按TE長度分類的TE類別的分布堆疊條形圖。
D. 對大于3.5 kb的TE進行LTR Copia預測結構域數量箱線圖,ns=不顯著。
E. 顯示大于3.5 kb的TE在基因組中分布示意圖。
F. 按TE長度分類的所有TEs或H3K27me3標記的TEs的TE超家族分布堆疊條形圖。
G. 顯示TE亞家族成員被H3K27me3標記的百分比與TE拷貝數量之間的相關性圖。
H. 基于每個TE拷貝的H3K27me3水平的熱圖。上圖:ATCOPIA12;下圖:ATCOPIA48。
(2)H3K27me3標記的TEs具有沉默且響應的基因表觀遺傳特征,其轉錄受PcG抑制
H3K27me3標記的TEs中有23%和22%分別含有H2A.Z或H2AK121ub標記,這些標記通常與動態轉錄抑制基因相關。這表明這些TEs具有沉默但可響應的基因特征。
在arp6(H2A.Z沉積缺失)和atbmi(H2AK121ub沉積缺失)突變體中,部分TEs的H3K27me3水平降低,表明H3K27me3的沉積依賴于H2A.Z和H2AK121ub。此外,在ref6 elf6 jmj13三重突變體中,許多H3K27me3標記的TEs顯示出更高的H3K27me3水平,表明這些TEs的H3K27me3模式受組蛋白去甲基化酶調控。
在swn clf PRC2雙突變體中,約2/3的H3K27me3標記的TEs顯著上調,表明H3K27me3在擬南芥中可以轉錄抑制TEs表達。
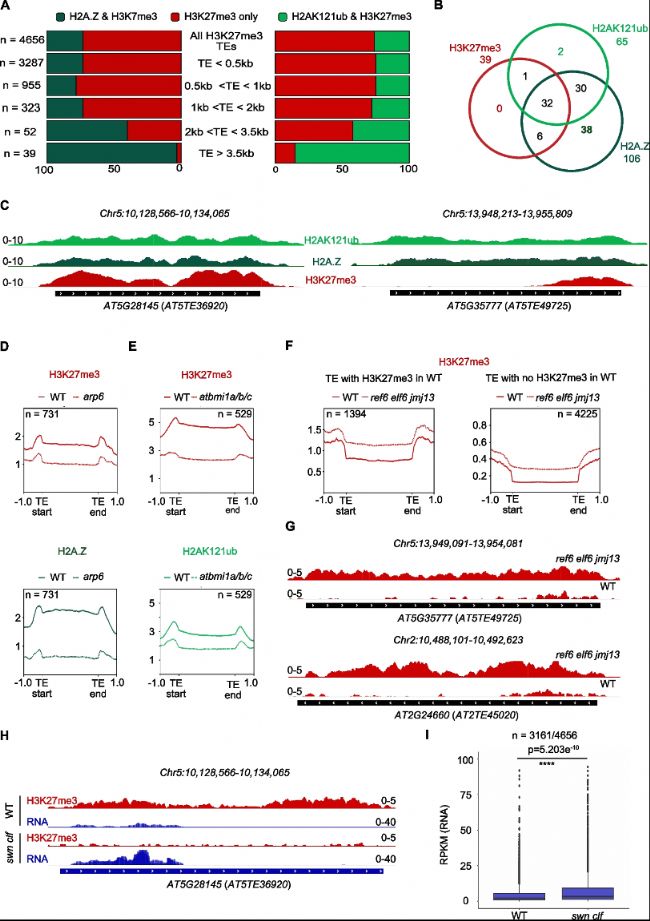
B. 維恩圖顯示在大于3.5 kb的TEs上H3K27me3、H2A.Z變體和H2AK121ub的存在情況。
C. 代表性基因組瀏覽器視圖展示來自第3類(被H3K27me3標記)的2個TEs的ChIP-seq數據,顯示了野生型(WT)中的H2AK121ub(淺綠色)、H2A.Z(深綠色)和H3K27me3(紅色)標記。
D. Metagenes圖顯示了在WT和arp6突變體(該突變體在H2A.Z摻入受影響的TE亞集中存在缺陷)中H3K27me3(上圖)和H2A.Z(下圖)的水平。
E. Metagenes圖顯示了在WT和atbmi1a/b/c突變體(該突變體在H2AK121ub沉積受影響的TE亞集中存在缺陷)中H3K27me3(上圖)和H2AK121ub(下圖)的水平。
F. Metagenes圖顯示了在WT和ref6 elf6 jmj13三重H3K27me3去甲基化酶突變體中H3K27me3的水平。左側圖表示在WT中已被H3K27me3標記但在ref6 elf6 jmj13中該標記增加的TEs。右側圖表示在WT中未被H3K27me3標記但在ref6 elf6 jmj13中該標記顯現的TEs。
G. 代表性基因組瀏覽器視圖展示了來自第3類的2個TEs在野生型和ref6 elf6 jmj13中的H3K27me3(紅色)標記。
H. 代表性瀏覽器視圖展示TE在野生型和swn clf雙突變體中的H3K27me3(紅色)和RNA(藍色)。
I. 箱線圖顯示與野生型相比,在swn clf雙突變體中顯著上調的第3類TEs(4656個中有3161個)。
(3)H3K27me3標記以順式決定方式被招募到新插入的轉基因TEs上
通過將三個不同的COPIA LTR逆轉錄轉座子序列克隆到二元載體中,并將其插入植物基因組,研究新插入的TEs是否能夠招募H3K27me3。結果表明,這些新插入的TEs能夠以序列特異性的方式招募H3K27me3,表明H3K27me3的招募依賴于TE序列本身的內在特征。
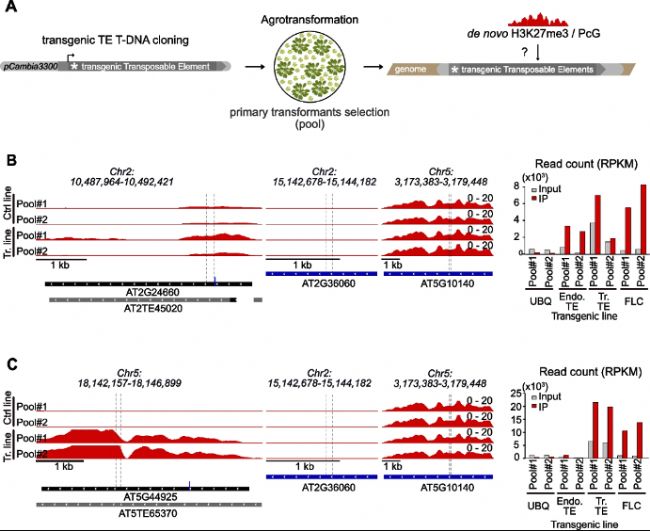
B. 左側圖:代表性基因組瀏覽器視圖顯示在兩個獨立的植物群體中,H3K27me3的平均分布(IP-INPUT)在COPIA12B序列(頂部圖)上,這些植物要么被轉化了無關的轉基因(Ctrl),要么被轉化了感興趣的TE。在“Ctrl”軌跡中的H3K27me3信號反映內源TE的H3K27me3狀態。在“Tr.”軌跡中的H3K27me3信號反映內源+轉基因的H3K27me3狀態:與對照線相比更高的信號表明除了內源基因外,轉基因TE上也招募了H3K27me3。轉基因群體之間的變異性反映了轉基因TE拷貝數的差異。中間圖:UBQ和FLC分別作為H3K27me3招募的陰性和陽性對照。右側圖:通過在引入SNP的區域對內源和轉基因COPIA12B序列進行H3K27me3免疫沉淀的定量來確定轉基因上的招募。
C. 左、中和右圖與B中的COPIA21序列相同。
(4)群體水平上的TE中在DNA甲基化和Polycomb標記之間轉換
通過比較擬南芥不同自然生態型(如Col-0和KBS-Mac-74)的H3K27me3和DNA甲基化數據,發現存在一類TEs(稱為“bifrons”),其在不同生態型中表現出表觀遺傳狀態的轉換,即從DNA甲基化到H3K27me3或反之。
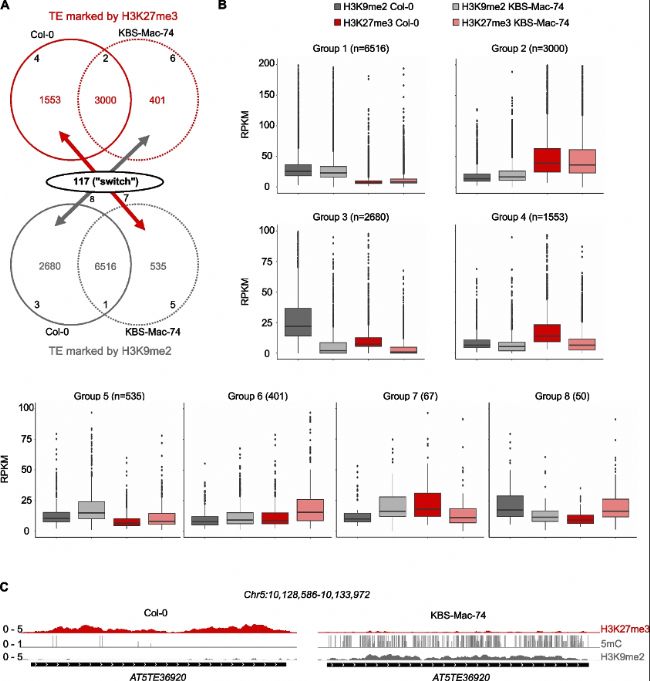
B. 盒須圖顯示在Col-0和KBS-Mac-74中,八個不同TE簇的H3K9me2水平(以RPKM表示)和H3K27me3水平。
C. 代表性基因組瀏覽器視圖顯示在Col-0和KBS-Mac-74兩個生態型中,一個同源TE拷貝的H3K27me3和DNA甲基化水平(紅色:H3K27me3,深灰色:CG甲基化,灰色:CHG甲基化,淺灰色:CHH甲基化,黑色條:TE注釋)。
(5)某些TEs上H3K27me3的沉積與TE的順式決定因子以及反式作用因子在遺傳上相關
通過對近500個擬南芥自然生態型的DNA甲基化數據進行全基因組關聯研究(GWAS),發現TEs的表觀遺傳狀態轉換與TE序列本身(cis-determinants)以及參與DNA甲基化的轉錄因子(trans factors)相關。
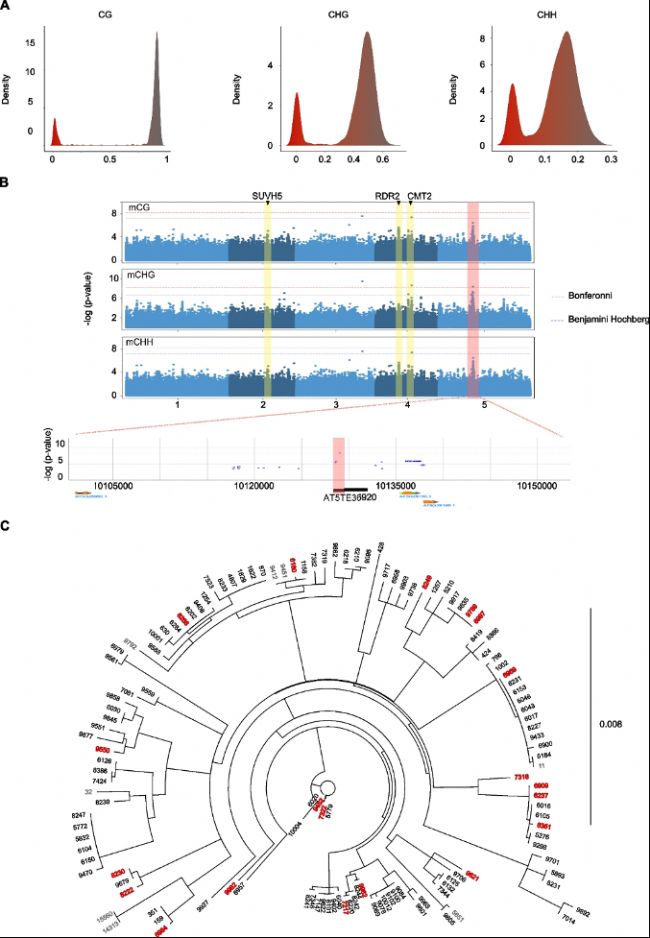
B. 曼哈頓圖展示了靶向COPIA12D(AT5TE36920)的單變量全基因組關聯研究(GWAS)結果,分別為mCG(上圖)、mCHG(中圖)和mCHH(下圖)。水平線表示全基因組顯著性水平。黃色框表示與DNA甲基化相關的反式作用因子的峰值,粉色框表示TE的順式區域。
C. 130個不同生態型中AT5TE36920序列的系統發育樹。右側標示了遺傳距離。紅色加粗的生態型表示COPIA12D無DNA甲基化,被認為被H3K27me3標記。黑色的生態型表示COPIA12D存在DNA甲基化。灰色的生態型表示沒有關于COPIA12D表觀遺傳狀態的信息。
易小結
本研究揭示了通過ChIP-seq等技術擬南芥中TEs的一種與H3K27me3相關的可變沉默機制。此外,研究還發現了TEs在物種水平上表觀遺傳狀態的動態轉換,這一現象可能擴展到其他多細胞真核生物。這些發現挑戰了傳統觀念,即擬南芥TEs僅通過DNA甲基化進行沉默,并為理解TEs在進化中的作用提供了新的視角。
ChIP-seq和BS測序技術在本研究中的作用
ChIP-seq技術:用于檢測H3K27me3在TEs上的分布情況,揭示了TEs的表觀遺傳特征以及H3K27me3與H2A.Z和H2Aub的共定位。通過ChIP-seq,研究人員鑒定出哪些TEs被H3K27me3標記,并分析這些標記的動態變化。
BS技術:用于分析TEs的DNA甲基化狀態,與ChIP-seq數據相結合,幫助研究人員理解TEs的表觀遺傳狀態轉換。通過亞硫酸鹽測序,研究人員能夠確定TEs在不同生態型中DNA甲基化差異,從而揭示表觀遺傳狀態轉換的分子基礎。
參考文獻:
Hure, V., Piron-Prunier, F., Yehouessi, T. et al. Alternative silencing states of transposable elements in Arabidopsis associated with H3K27me3. Genome Biol 26, 11 (2025). https://doi.org/10.1186/s13059-024-03466-6
近日,法國巴黎薩克雷大學細胞綜合生物學研究所Angélique Déléris團隊以模式生物擬南芥為研究對象,研究了植物中轉座元件(TEs)的表觀遺傳沉默機制,特別是與H3K27me3(組蛋白H3K27三甲基化)相關的沉默狀態。研究結果揭示了在野生型擬南芥中,許多TEs可以被H3K27me3單獨靶向并沉默(而不依賴DNA甲基化沉默機制)。此外,研究還發現了TEs在不同擬南芥自然生態型之間存在表觀遺傳狀態的轉換,即從DNA甲基化到H3K27me3的相互轉換,這一現象被稱為“bifrons”。相關研究成果以“Alternative silencing states of transposable elements in Arabidopsis associated with H3K27me3”為題發表于《Genome Biology》(IF:10.1)期刊。

標題:Alternative silencing states of transposable elements in Arabidopsis associated with H3K27me3(擬南芥中與 H3K27me3 相關的轉座因子可變沉默狀態)
發表時間:2025-01-20
期刊:Genome Biology
影響因子:IF10.1/Q1
技術平臺:ChIP-seq、BS
研究摘要
DNA/H3K9甲基化和Polycomb蛋白組(Polycomb-group,PcG)-H3K27me3沉默通路長期以來被認為在哺乳動物、植物和真菌中是互斥的,并且分別特異性地作用于轉座元件(TEs)和基因。但即使在DNA/H3K9甲基化機制缺失情況下,許多TEs仍然可以被H3K27me3靶向,有時甚至與DNA甲基化共存。
本研究結果揭示了在野生型擬南芥植物中,TEs也可以被H3K27me3單獨靶向并沉默。這些被H3K27me3標記的TEs不僅包括退化殘跡,還包括看似完整的拷貝,它們顯示出響應性PcG靶基因的表觀遺傳特征以及活躍的H3K27me3調控。同時,H3K27me3可以以TE特異性方式沉積在新插入的轉基因TE序列上,表明沉默由順式( cis)作用決定。最后通過比較擬南芥的自然生態型發現了一類TEs(稱為“bifrons”),它們的標記取決于生態型,要么被DNA甲基化標記,要么被H3K27me3標記。這種差異可以追溯到TEs的內在特征以及反式作用因子,并揭示TEs在其生命周期中表觀遺傳狀態變化。
總之,本研究揭示了開花植物中與H3K27me3相關的可變TE沉默模式,同時還表明了在物種水平上兩種表觀遺傳標記(DNA甲基化和H3K27me3)之間存在動態轉換,這一新的范式可能擴展到其他多細胞真核生物。
研究方法
(1)ChIP-seq技術:檢測H3K27me3在TEs上的分布情況。
(2)BS技術:用于分析TEs的DNA甲基化水平。
結果圖形
(1)在野生型擬南芥植物中,許多TE只由H3K27me3標記
研究發現,擬南芥基因組中有超過4000個TEs被H3K27me3標記,這些TEs不僅包括退化的殘跡,還包括看似完整的拷貝。這些TEs顯示出與響應環境或發育信號的經典PcG靶基因相似的染色質特征,其H3K27me3模式受到組蛋白H3K27去甲基化酶的積極調控。
根據H3K27me3和DNA甲基化的覆蓋長度,將TEs分為四類。其中,“class 3”僅被H3K27me3標記,占TE總數的15%,但僅占TE堿基的8.4%。這些TEs傾向于分布在短TE類別中(尤其是小于1kb的TE),而長TEs(大于3.5kb)通常與DNA甲基化相關,盡管一小部分長TEs(大于3.5kb)僅被H3K27me3標記。
這些TEs在基因組中的分布與DNA甲基化的TEs不同,它們不僅存在于異染色質區域,還分散在常染色質臂和近著絲粒區域。

圖1:在野生型擬南芥(Col-0)中,許多轉座元件(TEs)被H3K27me3標記。
B. 基于表觀遺傳修飾的TE分類的維恩圖。當CG甲基化在TE長度上的平均值>50%時,TE被定義為“DNA甲基化”(類別1-2)。當H3K27me3 peaks至少覆蓋TE長度的20%時,TE被定義為處于H3K27me3狀態。
C. 按TE長度分類的TE類別的分布堆疊條形圖。
D. 對大于3.5 kb的TE進行LTR Copia預測結構域數量箱線圖,ns=不顯著。
E. 顯示大于3.5 kb的TE在基因組中分布示意圖。
F. 按TE長度分類的所有TEs或H3K27me3標記的TEs的TE超家族分布堆疊條形圖。
G. 顯示TE亞家族成員被H3K27me3標記的百分比與TE拷貝數量之間的相關性圖。
H. 基于每個TE拷貝的H3K27me3水平的熱圖。上圖:ATCOPIA12;下圖:ATCOPIA48。
(2)H3K27me3標記的TEs具有沉默且響應的基因表觀遺傳特征,其轉錄受PcG抑制
H3K27me3標記的TEs中有23%和22%分別含有H2A.Z或H2AK121ub標記,這些標記通常與動態轉錄抑制基因相關。這表明這些TEs具有沉默但可響應的基因特征。
在arp6(H2A.Z沉積缺失)和atbmi(H2AK121ub沉積缺失)突變體中,部分TEs的H3K27me3水平降低,表明H3K27me3的沉積依賴于H2A.Z和H2AK121ub。此外,在ref6 elf6 jmj13三重突變體中,許多H3K27me3標記的TEs顯示出更高的H3K27me3水平,表明這些TEs的H3K27me3模式受組蛋白去甲基化酶調控。
在swn clf PRC2雙突變體中,約2/3的H3K27me3標記的TEs顯著上調,表明H3K27me3在擬南芥中可以轉錄抑制TEs表達。

圖2:H3K27me3在TEs上可與H2AUb和H2A.Z相關聯,表現出活躍的去甲基化和轉錄抑制。
A. 堆疊條形圖顯示在H3K27me3標記的TEs中,按大小范圍分類的被H2A.Z變體或H2AK121泛素化標記TEs的分布情況。如果一個TE同時存在H3K27me3和H2A.Z或H3K27me3和H2AK121ub的峰重疊,則該TE被認為共標記。B. 維恩圖顯示在大于3.5 kb的TEs上H3K27me3、H2A.Z變體和H2AK121ub的存在情況。
C. 代表性基因組瀏覽器視圖展示來自第3類(被H3K27me3標記)的2個TEs的ChIP-seq數據,顯示了野生型(WT)中的H2AK121ub(淺綠色)、H2A.Z(深綠色)和H3K27me3(紅色)標記。
D. Metagenes圖顯示了在WT和arp6突變體(該突變體在H2A.Z摻入受影響的TE亞集中存在缺陷)中H3K27me3(上圖)和H2A.Z(下圖)的水平。
E. Metagenes圖顯示了在WT和atbmi1a/b/c突變體(該突變體在H2AK121ub沉積受影響的TE亞集中存在缺陷)中H3K27me3(上圖)和H2AK121ub(下圖)的水平。
F. Metagenes圖顯示了在WT和ref6 elf6 jmj13三重H3K27me3去甲基化酶突變體中H3K27me3的水平。左側圖表示在WT中已被H3K27me3標記但在ref6 elf6 jmj13中該標記增加的TEs。右側圖表示在WT中未被H3K27me3標記但在ref6 elf6 jmj13中該標記顯現的TEs。
G. 代表性基因組瀏覽器視圖展示了來自第3類的2個TEs在野生型和ref6 elf6 jmj13中的H3K27me3(紅色)標記。
H. 代表性瀏覽器視圖展示TE在野生型和swn clf雙突變體中的H3K27me3(紅色)和RNA(藍色)。
I. 箱線圖顯示與野生型相比,在swn clf雙突變體中顯著上調的第3類TEs(4656個中有3161個)。
(3)H3K27me3標記以順式決定方式被招募到新插入的轉基因TEs上
通過將三個不同的COPIA LTR逆轉錄轉座子序列克隆到二元載體中,并將其插入植物基因組,研究新插入的TEs是否能夠招募H3K27me3。結果表明,這些新插入的TEs能夠以序列特異性的方式招募H3K27me3,表明H3K27me3的招募依賴于TE序列本身的內在特征。

圖3:新插入的TE序列可以以一種指導性的方式從頭招募H3K27me3。
A. 實驗方案:將單個TE序列克隆到二元載體中,將其引入植物基因組,并在T1代的大規模植株群體(15-20株)中通過H3K27me3 ChIP-seq進行研究,以稀釋位置效應。B. 左側圖:代表性基因組瀏覽器視圖顯示在兩個獨立的植物群體中,H3K27me3的平均分布(IP-INPUT)在COPIA12B序列(頂部圖)上,這些植物要么被轉化了無關的轉基因(Ctrl),要么被轉化了感興趣的TE。在“Ctrl”軌跡中的H3K27me3信號反映內源TE的H3K27me3狀態。在“Tr.”軌跡中的H3K27me3信號反映內源+轉基因的H3K27me3狀態:與對照線相比更高的信號表明除了內源基因外,轉基因TE上也招募了H3K27me3。轉基因群體之間的變異性反映了轉基因TE拷貝數的差異。中間圖:UBQ和FLC分別作為H3K27me3招募的陰性和陽性對照。右側圖:通過在引入SNP的區域對內源和轉基因COPIA12B序列進行H3K27me3免疫沉淀的定量來確定轉基因上的招募。
C. 左、中和右圖與B中的COPIA21序列相同。
(4)群體水平上的TE中在DNA甲基化和Polycomb標記之間轉換
通過比較擬南芥不同自然生態型(如Col-0和KBS-Mac-74)的H3K27me3和DNA甲基化數據,發現存在一類TEs(稱為“bifrons”),其在不同生態型中表現出表觀遺傳狀態的轉換,即從DNA甲基化到H3K27me3或反之。

圖4:通過不同生態型之間的比較,可視化TEs上沉默表觀遺傳標記之間的轉換。
A. 維恩圖顯示在兩種不同生態型(Col-0和KBS-Mac-74)中,被H3K27me3標記的TEs(頂部,紅色數字)和被DNA甲基化標記的TEs(底部,灰色數字)。兩個維恩圖的交集顯示了在一種生態型中被H3K27me3標記而在另一種生態型中被H3K9me2標記的TEs(107個)。B. 盒須圖顯示在Col-0和KBS-Mac-74中,八個不同TE簇的H3K9me2水平(以RPKM表示)和H3K27me3水平。
C. 代表性基因組瀏覽器視圖顯示在Col-0和KBS-Mac-74兩個生態型中,一個同源TE拷貝的H3K27me3和DNA甲基化水平(紅色:H3K27me3,深灰色:CG甲基化,灰色:CHG甲基化,淺灰色:CHH甲基化,黑色條:TE注釋)。
(5)某些TEs上H3K27me3的沉積與TE的順式決定因子以及反式作用因子在遺傳上相關
通過對近500個擬南芥自然生態型的DNA甲基化數據進行全基因組關聯研究(GWAS),發現TEs的表觀遺傳狀態轉換與TE序列本身(cis-determinants)以及參與DNA甲基化的轉錄因子(trans factors)相關。

圖5:表觀遺傳轉換的遺傳決定因子。
A. 圖表顯示了一個同源TE,即COPIA12D(AT5TE36920)的DNA甲基化值,以及在三個序列環境中(CG、CHG和CHH)的甲基化密度分布情況。B. 曼哈頓圖展示了靶向COPIA12D(AT5TE36920)的單變量全基因組關聯研究(GWAS)結果,分別為mCG(上圖)、mCHG(中圖)和mCHH(下圖)。水平線表示全基因組顯著性水平。黃色框表示與DNA甲基化相關的反式作用因子的峰值,粉色框表示TE的順式區域。
C. 130個不同生態型中AT5TE36920序列的系統發育樹。右側標示了遺傳距離。紅色加粗的生態型表示COPIA12D無DNA甲基化,被認為被H3K27me3標記。黑色的生態型表示COPIA12D存在DNA甲基化。灰色的生態型表示沒有關于COPIA12D表觀遺傳狀態的信息。
易小結
本研究揭示了通過ChIP-seq等技術擬南芥中TEs的一種與H3K27me3相關的可變沉默機制。此外,研究還發現了TEs在物種水平上表觀遺傳狀態的動態轉換,這一現象可能擴展到其他多細胞真核生物。這些發現挑戰了傳統觀念,即擬南芥TEs僅通過DNA甲基化進行沉默,并為理解TEs在進化中的作用提供了新的視角。
ChIP-seq和BS測序技術在本研究中的作用
ChIP-seq技術:用于檢測H3K27me3在TEs上的分布情況,揭示了TEs的表觀遺傳特征以及H3K27me3與H2A.Z和H2Aub的共定位。通過ChIP-seq,研究人員鑒定出哪些TEs被H3K27me3標記,并分析這些標記的動態變化。
BS技術:用于分析TEs的DNA甲基化狀態,與ChIP-seq數據相結合,幫助研究人員理解TEs的表觀遺傳狀態轉換。通過亞硫酸鹽測序,研究人員能夠確定TEs在不同生態型中DNA甲基化差異,從而揭示表觀遺傳狀態轉換的分子基礎。
參考文獻:
Hure, V., Piron-Prunier, F., Yehouessi, T. et al. Alternative silencing states of transposable elements in Arabidopsis associated with H3K27me3. Genome Biol 26, 11 (2025). https://doi.org/10.1186/s13059-024-03466-6
標簽:
DNA甲基化
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com





