“Range+T”模式,助力Pacbio HiFi測序高質量文庫構建
Pippin 片段回收儀增加全新“Range+T”模式,助力Pacbio HiFi測序高質量文庫構建
PacBio 獨有的HiFi測序技術,可同時實現長讀長和高準確性的DNA測序,在解析復雜的基因組區域、檢測結構變異、揭示罕見變異和研究表觀遺傳學等多個領域有廣泛應用。為了確保HiFi測序的產量和質量,需要將DNA文庫大小控制在15-18kb范圍內。因此,高精度的片段篩選是PacBio HiFi測序的關鍵步驟之一。Sage Science公司提供的Pippin系列全自動DNA片段回收儀--基于脈沖場電泳,可以實現上述需求,現已成為HiFi文庫構建的金標準和建庫必備工具之一。
為了獲得更高質量的HiFi文庫,Sage Science公司不斷優化篩選方法,并于今年6月推出了全新的長片段回收模式“Range+T”。結合相對應的預制膠試劑盒,“Range+T”模式可有效提高9-30kb范圍內片段篩選的分辨率,協助用戶更精準的控制HiFi文庫大小,實現片段讀取長度和質量間的平衡,得到更好的基因組組裝結果。
運用“Range+T”模式,只需設置回收范圍起點DNA的bp值,以及回收時間(一般5-30min)即可,例如:15kb+10min。此模式的優點包括:(1)有效去除設置起點外的小片段DNA;(2)可以更精準的控制文庫回收的上限;(3)可以更精準調整文庫分布的寬度。近日,Sage Science公司攜手亞利桑那大學基因組研究所,以植物基因組為樣本,共同測試了“Range+T”模式對于增加PacBio HiFi文庫平均讀長的能力。由于植物基因組的復雜性更高(多倍體、重復序列、大小差異大等),因此更長的HiFi讀取長度對植物基因組研究更重要。
為了獲得更高質量的HiFi文庫,Sage Science公司不斷優化篩選方法,并于今年6月推出了全新的長片段回收模式“Range+T”。結合相對應的預制膠試劑盒,“Range+T”模式可有效提高9-30kb范圍內片段篩選的分辨率,協助用戶更精準的控制HiFi文庫大小,實現片段讀取長度和質量間的平衡,得到更好的基因組組裝結果。
運用“Range+T”模式,只需設置回收范圍起點DNA的bp值,以及回收時間(一般5-30min)即可,例如:15kb+10min。此模式的優點包括:(1)有效去除設置起點外的小片段DNA;(2)可以更精準的控制文庫回收的上限;(3)可以更精準調整文庫分布的寬度。近日,Sage Science公司攜手亞利桑那大學基因組研究所,以植物基因組為樣本,共同測試了“Range+T”模式對于增加PacBio HiFi文庫平均讀長的能力。由于植物基因組的復雜性更高(多倍體、重復序列、大小差異大等),因此更長的HiFi讀取長度對植物基因組研究更重要。

“Range+T”片段篩選模式增加PacBio HiFi平均讀長
研究成果:
1. 不同回收時間對回收得率和回收范圍的影響
小麥基因組DNA經打斷儀處理后,使用PippinHT回收目標片段。使用“Range+T”回收模式,不同樣本的回收起點均設置為15kb,回收時間分別設置為13-30min。
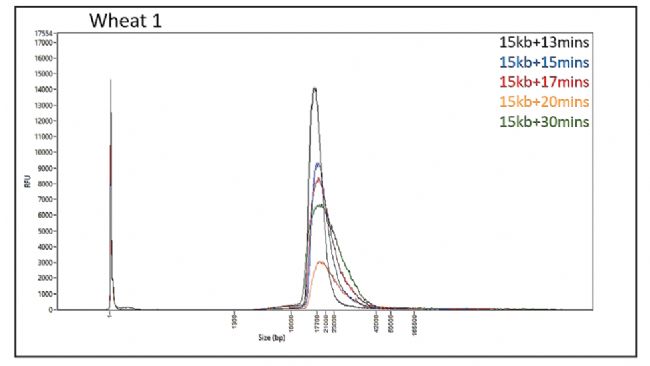
圖1“Range+T”模式回收的精確性
回收后檢測結果顯示,不同泳道回收起點的位置相同,回收的終點與設置的回收時間長短相關,設置30min的回收時間可以回收樣品中更多的HWM DNA。
2. 小片段去除效果
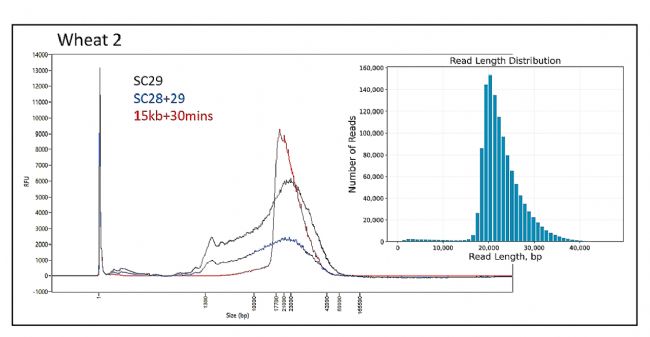
圖2“Range+T”模式小片段去除結果
如圖2所示,SC28+29樣品混合后(藍色),PippinHT “Range+T”模式設置為13kb+30min進行片段回收,回收后樣品有效去除回收起點以下的小片段DNA(紅色),結果也在Pacbio測序結果中reads長度分布圖(圖2中測序結果圖)得以體現。
3. 重復性測試
8個木薯樣本的基因組DNA被打斷成13-25kb(平均長度18kb),“Range+T”模式設置為13kb+30min進行片段回收,回收得率在33-45%之間,回收后平均片段長度在19-20kb之間,HiFi reads結果長度保持一致。
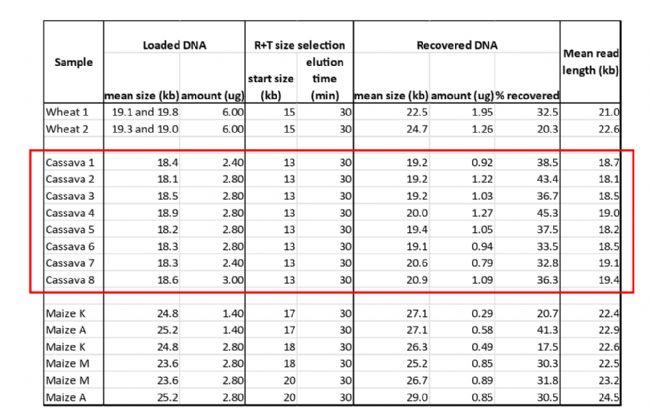
表1 “Range+T”模式重復性測試結果
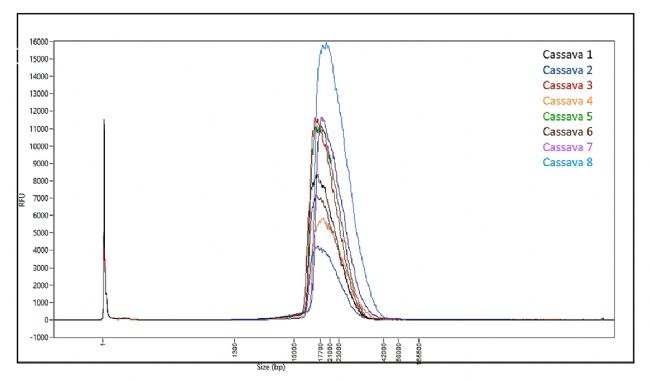
圖3 “Range+T”模式重復性測試Femto Pulse檢測結果
4. 長片段回收能力
以3個玉米樣本為例,基因組DNA打斷成15-35kb片段(平均長度23-25kb)。“Range+T”模式回收起點分別設置在17kb-20kb,回收時間均為30min。結果表明DNA回收得率從17%到41%不等,可檢測的最大片段長度約為50kb,提高回收起點會得到更大的片段分布,從而產生更長的平均HiFi read長度。
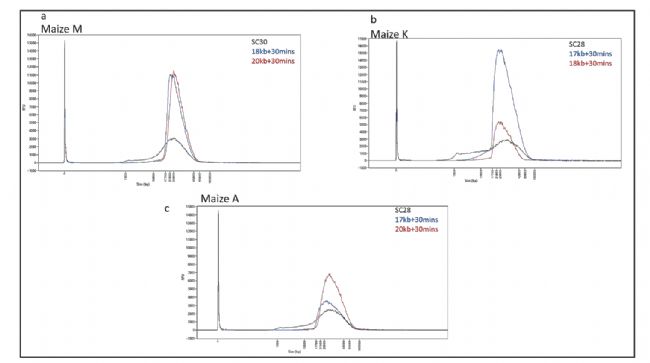
圖4 “Range+T”模式長片段回收測試結果
5. 對測序質量影響
為了研究測序結果在產量、read質量和平均HiFi read長度之間得到更好的平衡,作者對比了PippinHT 之前軟件版本9-13kb篩選模式的測序結果(圖5中的藍色符號)與最新“Range+T” 模式的測序結果(圖5中的橙色符號)。使用“Range+T”模式,回收起點設置為15kb,其產生的reads平均長度在21-22kb以上,且30-40kb長度reads數量有顯著提高。更重要的是,數據(圖5a)表明在13kb至22kb的范圍內,隨著平均HiFi read長度的增加,HiFi測序數據產量幾乎沒有下降。由于PacBio測序中聚合酶讀取長度有限,短的SMRTbell文庫比長文庫具有更高的QV分數,但此次實驗中較長文庫的最低平均QV分數仍然相當好,為28。88%的較長文庫(橙色符號)的平均QV得分在30以上(圖5.b-c)。

圖5 不同設置HiFi測序產量及QV結果
為了更好地說明較長文庫的質量結果,圖6顯示了一個平均subreads長度為22.6kb的玉米樣本的reads質量分布,其中約40%的reads得分在Q30以上。
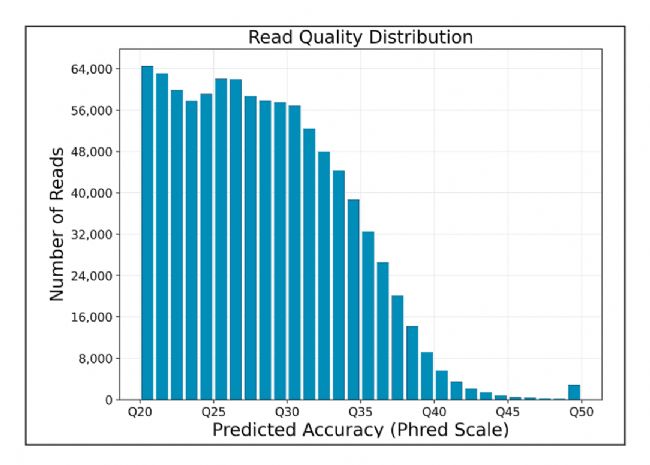
圖6 玉米文庫Read質量分布結果,平均長度為22.6kb
為了體現更長read對基因組組裝的影響,作者組裝了三個數據集。數據來自基于“Range+T”模式的PacBio-HiFi文庫(表1的玉米樣本)。文庫的覆蓋率為18×,平均HiFi read長度為20.6kb。生成了以下三個具有不同特征的數據集(表2):
- “Small”數據集:從所有reads的3'端刪除4000個bp。這使得reads保留了原始分布形狀,但reads長度整體被縮短,平均長度為16kb。
- “Long”數據集:將原始的20kb文庫測序覆蓋率調整為~14.5×,以匹配“Small”數據集的數據量。
- “Narrow”數據集:所有最長reads被截斷至約17kb,用以模擬窄分布的SMRTbell文庫。為了與其他兩個數據集相匹配,總體覆蓋率也有所降低。
“Small”數據集代表PacBio文庫制備的當前標準,而“Long”數據集結果證明了“Range+T”模式的優勢。與其他數據集相比,“Long”數據集具有最低的contigs數量、最高的平均contigs長度和最高的Nx值(表2)。此外,開發“Narrow”數據集是為了研究更長read對改進組裝效果的貢獻。
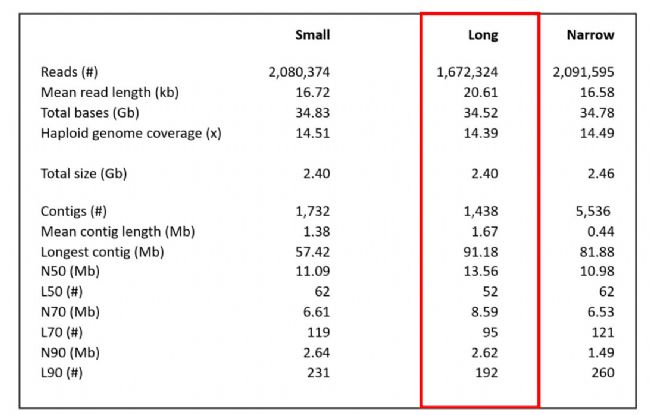
表2 玉米M組裝統計數據集
結論:
測試結果說明PippinHT最新的“Range+T”模式,可以更加靈活地選擇PacBio HiFi文庫的大小。“Range+T”模式具備較好的重復性,可以有效去除短片段,且使平均read長度超過當前標準。此外,新模式可以控制文庫片段范圍的上限,更長的文庫并沒有影響測序質量和產量。與較小的HiFi文庫相比,更有助于大而復雜基因組的連續組裝。
目前PippinHT “Range+T”模式的新版軟件已經發布,Blue Pippin也將在近期發布,詳細信息請聯系:
▼環亞生物科技有限公司▼
地址:上海市閔行區友東路358號閔欣大廈1號樓701/703/705室
電話:021-54583565
郵箱:info@apgbio.com
網址:www.apgbio.com
Sage Science是全球領先的研發制造Pippin系列全自動核酸電泳片段回收儀的生產廠家,擁有美國技術專利,各個型號產品可以高效完成不同長度范圍的DNA片段的精準回收,廣泛用于二代測序、三代測序以及多種分子生物學相關領域。
環亞生物科技有限公司作為美國Sage Science公司在中國區的獨家代理商,自2011年以來將Sage Science的產品線引入國內,一直為國內用戶提供專業的全自動核酸片段回收系統的安裝測試、應用培訓,技術支持與售后維護工作,贏得客戶的一致好評與信任。環亞生物科技有限公司將一如既往的支持越來越多的Sage Science用戶。
Copyright(C) 1998-2025 生物器材網 電話:021-64166852;13621656896 E-mail:info@bio-equip.com





