AI人工智能在蛋白質(zhì)結(jié)構(gòu)預(yù)測、功能預(yù)測及設(shè)計中的作用
在生物技術(shù)的前沿領(lǐng)域,AI 正在掀起一場蛋白質(zhì)研究的革命。AI 幫助科學(xué)家以前所未有的精度預(yù)測蛋白質(zhì)結(jié)構(gòu)和功能、從頭設(shè)計新型蛋白質(zhì)、解開生命分子層面的奧秘。蛋白質(zhì)生產(chǎn)技術(shù)的進步,如 AlphaFold 和人工智能驅(qū)動技術(shù),不僅改寫了蛋白質(zhì)研究的規(guī)則,也為從藥物開發(fā)到疾病診斷和合成生物學(xué)等各個領(lǐng)域帶來了許多可能性。
人工智能在蛋白質(zhì)結(jié)構(gòu)預(yù)測中的作用
蛋白質(zhì)研究一直是生物技術(shù)領(lǐng)域中的核心難題,其復(fù)雜性源于蛋白質(zhì)多樣的三維結(jié)構(gòu)和動態(tài)特性。長期以來,科學(xué)家們一直使用X射線晶體學(xué)、核磁共振(NMR)和冷凍電子顯微鏡等傳統(tǒng)工具來解析蛋白質(zhì)結(jié)構(gòu)。然而,這些方法往往成本高昂、耗時,并對特定蛋白質(zhì)類型的解析存在局限性。這些傳統(tǒng)技術(shù)在應(yīng)對蛋白質(zhì)的動態(tài)性和環(huán)境依賴性方面也有較大挑戰(zhàn),導(dǎo)致實際解析的結(jié)構(gòu)可能無法準確反映蛋白質(zhì)在生理條件下的狀態(tài)。科學(xué)家們亟需新的工具,能夠更快速、高效、低成本地完成復(fù)雜蛋白質(zhì)結(jié)構(gòu)的預(yù)測與解析,以滿足現(xiàn)代生命科學(xué)研究的需求。
AI如何突破蛋白質(zhì)研究困境?
隨著計算機技術(shù)和算法的不斷進步,科學(xué)家們開始探索使用人工智能和機器學(xué)習(xí)方法來解決蛋白質(zhì)結(jié)構(gòu)預(yù)測問題。
2020年,DeepMind團隊推出了突破性工具AlphaFold,并經(jīng)歷了不斷的發(fā)展和完善。該模型利用深度學(xué)習(xí)精確預(yù)測蛋白質(zhì)三維結(jié)構(gòu),顯著提升了蛋白質(zhì)結(jié)構(gòu)預(yù)測的準確性和速度,為生物學(xué)研究帶來了革命性進展。AlphaFold 使用深度學(xué)習(xí)模型,通過分析多序列比對 (MSA) 中的進化協(xié)變數(shù)據(jù),預(yù)測氨基酸殘基之間的距離,從而揭示蛋白質(zhì)結(jié)構(gòu)的空間構(gòu)造。神經(jīng)網(wǎng)絡(luò)會預(yù)測一個 “距離圖 ”或殘基-殘基距離的概率圖,為折疊過程提供指導(dǎo)。利用這些距離信息,該模型會進行優(yōu)化(如梯度下降),以確定蛋白質(zhì)的最終三維結(jié)構(gòu)。
2020年,AlphaFold 2.0在蛋白質(zhì)結(jié)構(gòu)預(yù)測競賽CASP14上一鳴驚人,獲得了98.5%的蛋白質(zhì)結(jié)構(gòu)預(yù)測率。作為 Alphafold 的升級版本,它能在幾分鐘內(nèi)預(yù)測出典型蛋白質(zhì)的結(jié)構(gòu),精度通常在1 Å 以內(nèi),接近碳原子寬度(約1.4 Å)。這種精確度代表了計算生物學(xué)的重大飛躍,為了解蛋白質(zhì)折疊的復(fù)雜性提供了強有力的工具。
AlphaFold 2.0 引入了一種名為 Evoformer 的先進神經(jīng)網(wǎng)絡(luò)架構(gòu)。該架構(gòu)利用進化和空間關(guān)系處理多序列比對(MSA)和成對殘基信息。通過三角更新和注意力機制,幫助模型捕捉遠程依賴性和空間關(guān)系,這對準確預(yù)測蛋白質(zhì)折疊至關(guān)重要。它還包括一個結(jié)構(gòu)模塊,直接預(yù)測三維原子坐標,并采用循環(huán)機制進行迭代優(yōu)化。通過端到端訓(xùn)練和獨特的自蒸餾過程,AlphaFold 2達到了接近實驗的精度,能夠預(yù)測以前具有挑戰(zhàn)性的復(fù)雜和新型結(jié)構(gòu)。
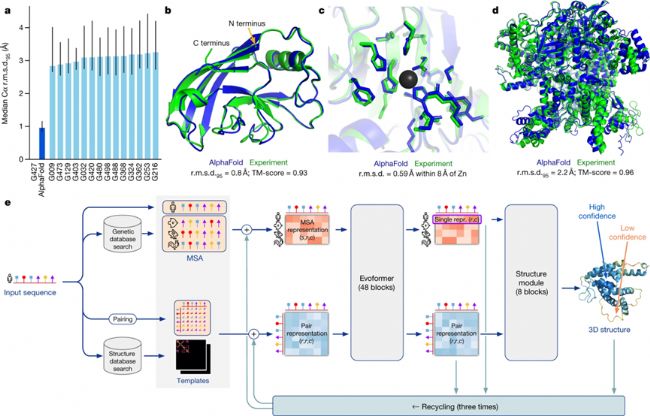
AlphaFold 2.0 模型架構(gòu)
2021年, David Baker的團隊推出了革命性工具RoseTTAFold。這一開源蛋白質(zhì)結(jié)構(gòu)預(yù)測工具采用了獨特的三軌網(wǎng)絡(luò)架構(gòu),能夠同時處理序列、距離和坐標信息,使得預(yù)測的精準度和速度顯著提高。
- 序列軌道:處理氨基酸序列信息。
- 距離軌道:處理氨基酸對之間相互作用的信息。
- 坐標軌道:處理局部結(jié)構(gòu)特征,如二級結(jié)構(gòu)和溶劑可及性。
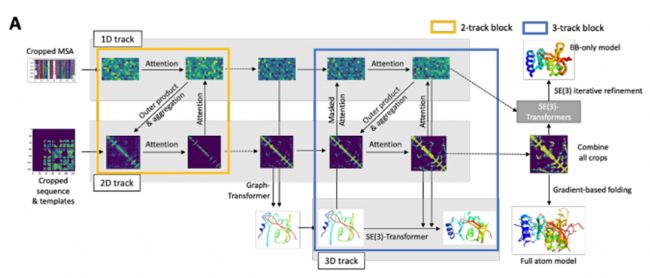
RoseTTAFold 的三維軌道架構(gòu)
該模型在不同通道中同時處理序列、距離和坐標信息,通過通道間的信息不斷交換來迭代完善蛋白質(zhì)結(jié)構(gòu)。RoseTTAFold的設(shè)計實現(xiàn)了一種多任務(wù)學(xué)習(xí)方法,可以同時優(yōu)化多個相關(guān)任務(wù),如距離圖預(yù)測、角度圖預(yù)測和接觸圖預(yù)測,有助于提高整體預(yù)測精度。
人工智能在蛋白質(zhì)功能預(yù)測中的作用
蛋白質(zhì)的功能是在基因本體(GO)中被定義的,其依據(jù)分子功能(MFO)、在生物過程里的作用(BPO)以及在細胞成分中的位置(CCO)來對蛋白質(zhì)實施分類。借助對同源蛋白質(zhì)的注釋,諸如UniProtKB/Swiss - Prot這類數(shù)據(jù)庫,為數(shù)千種生物以及超過55萬種蛋白質(zhì)提供了經(jīng)過整理的GO數(shù)據(jù)。
然而,數(shù)據(jù)庫中大部分蛋白質(zhì)缺少功能注釋,現(xiàn)有的注釋大多源于耗時的實驗。基于AI的預(yù)測方法,融合了氨基酸序列、結(jié)構(gòu)信息以及蛋白質(zhì)-蛋白質(zhì)相互作用(PPI)網(wǎng)絡(luò),使得蛋白質(zhì)功能預(yù)測更加高效,顯著提高了預(yù)測準確性和速度。這為填補蛋白質(zhì)功能注釋的空白提供了可擴展的解決方案。通過運用深度學(xué)習(xí)和文獻知識,這些工具能夠更高效且更精準地進行功能預(yù)測,從而加深我們對蛋白質(zhì)在健康和疾病中所起作用的理解。
DeepGO是第一個基于深度學(xué)習(xí)的預(yù)測模型,它通過將深度學(xué)習(xí)應(yīng)用于蛋白質(zhì)序列和相互作用數(shù)據(jù)來預(yù)測蛋白質(zhì)功能。該模型以蛋白質(zhì)的氨基酸序列作為輸入,使用卷積神經(jīng)網(wǎng)絡(luò)(CNNs)從中提取特征。DeepGO還結(jié)合了蛋白質(zhì)-蛋白質(zhì)相互作用(PPI)網(wǎng)絡(luò),使模型能夠利用蛋白質(zhì)之間的功能關(guān)系。通過這種方法,DeepGO分配基因本體(GO)術(shù)語,根據(jù)分子功能、生物過程和細胞成分對蛋白質(zhì)功能進行分類,這對全面的蛋白質(zhì)功能預(yù)測很有效果。
DeepGO-SE是一種用于蛋白質(zhì)功能預(yù)測的高級模型,它通過一種稱為近似語義蘊含的過程,將基因本體 (GO) 中的知識納入其中。該模型的運行分為三個關(guān)鍵步驟:
1. 構(gòu)建近似模型:利用GO的公理和蛋白質(zhì)功能斷言創(chuàng)建近似模型,其中ELEmbeddings以幾何表示法捕捉 GO 中的語義關(guān)系。
2. 蛋白質(zhì)嵌入和優(yōu)化:蛋白質(zhì)序列用預(yù)訓(xùn)練的ESM2模型的嵌入表示。然后在近似模型中對這些嵌入進行定位,以最大限度地提高 “蛋白質(zhì)具有C功能 ”這樣的語句的可能性,從而指導(dǎo)精確的功能預(yù)測。
3. 多模型聚合:重復(fù)這個過程以生成多個模型,最終預(yù)測基于所有模型中都成立的真值,有效地捕捉蘊含關(guān)系。
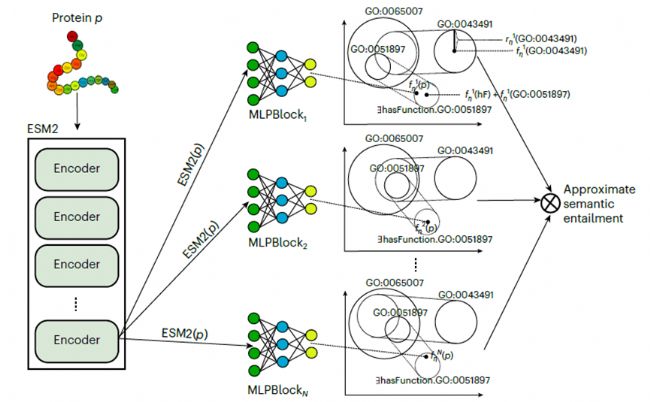 DeepGO-SE模型
DeepGO-SE模型
蛋白質(zhì)設(shè)計已經(jīng)有了顯著的發(fā)展,從最初通過PCR的誘變引入特定突變來調(diào)控蛋白質(zhì)結(jié)構(gòu),到應(yīng)用先進計算方法構(gòu)建具有所需特性的新型蛋白質(zhì)。如今,隨著結(jié)構(gòu)生物學(xué)、計算建模的快速發(fā)展,AI蛋白質(zhì)設(shè)計比以往任何時候都更加精確,也更容易獲得。AI驅(qū)動的設(shè)計技術(shù)既能優(yōu)化蛋白質(zhì),增強蛋白質(zhì)的天然功能(如親和力和穩(wěn)定性),還具備從頭構(gòu)建全新蛋白質(zhì)的能力,以實現(xiàn)特定的功能、結(jié)構(gòu)和應(yīng)用,為藥物發(fā)現(xiàn)、工業(yè)酶工程等領(lǐng)域帶來前所未有的創(chuàng)新可能性。
ProteinMPNN是一種用于高效蛋白質(zhì)序列設(shè)計的深度學(xué)習(xí)模型,它繞過了傳統(tǒng)基于物理的方法(如Rosetta)的計算需求。通過直接從結(jié)構(gòu)數(shù)據(jù)中學(xué)習(xí)并編碼空間關(guān)系, ProteinMPNN 能準確預(yù)測折疊成目標結(jié)構(gòu)的序列。該模型在設(shè)計復(fù)雜的組裝(如四面體納米粒子)和保持結(jié)合親和力(即使發(fā)生點突變)方面表現(xiàn)出色。該模型能夠創(chuàng)建各種類型的蛋白質(zhì),包括單體、組裝體和納米顆粒,是提高蛋白質(zhì)溶解度、穩(wěn)定性和功能性的強大工具。
RFdiffusion采用創(chuàng)新方法,利用擴散模型,通過迭代去噪,將蛋白質(zhì)骨架從最初的噪音細化為現(xiàn)實結(jié)構(gòu)。通過結(jié)合特定的結(jié)構(gòu)基序,它創(chuàng)造出多樣化、復(fù)雜的蛋白質(zhì),以滿足特定需求,如對稱組裝體和功能基序。RFdiffusion在生成新型拓撲結(jié)構(gòu)方面的精確性使其能夠應(yīng)用于前沿領(lǐng)域,從治療支架到復(fù)雜結(jié)構(gòu)設(shè)計。
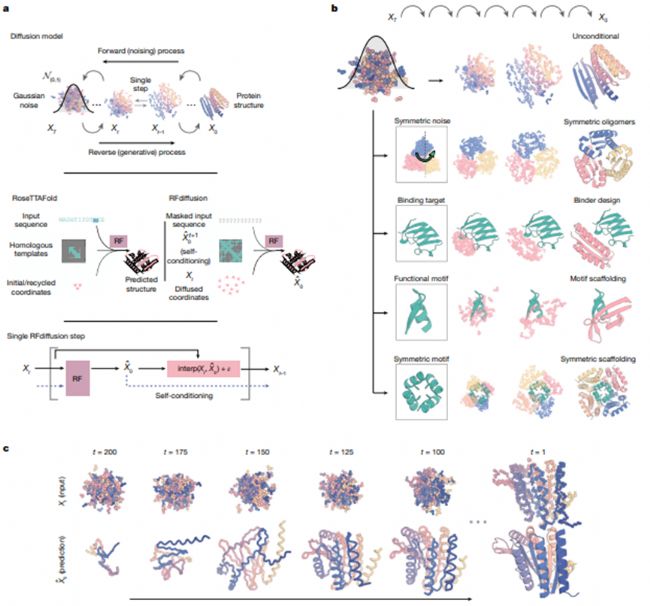 利用RFdiffusion設(shè)計蛋白質(zhì)
利用RFdiffusion設(shè)計蛋白質(zhì)
ProGen借鑒NLP的語言模型技術(shù),生成反映進化模式和生化特性的蛋白質(zhì)序列,從而能夠創(chuàng)建具有特定功能的序列。ProGen使用轉(zhuǎn)換器來模擬驅(qū)動穩(wěn)定性和功能性的序列特征,使其高度適用于各種應(yīng)用,從酶設(shè)計到生成具有特定結(jié)合親和力的序列。
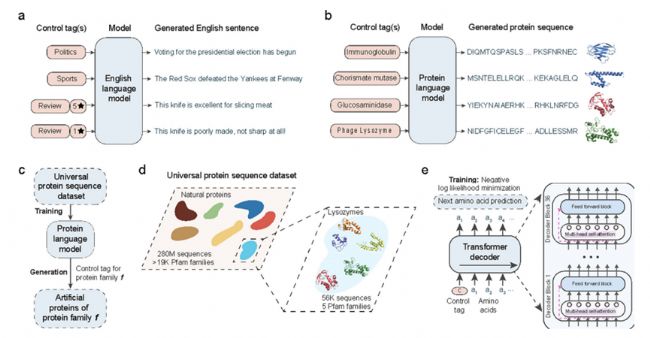 利用條件語言建模生成人工蛋白質(zhì)
利用條件語言建模生成人工蛋白質(zhì)
AlphaProteo專注于創(chuàng)建高親和力蛋白質(zhì)結(jié)合劑,利用結(jié)構(gòu)引導(dǎo)序列生成技術(shù)開發(fā)針對精確蛋白質(zhì)位點的結(jié)合劑。通過將生成模型與高級過濾器相結(jié)合。AlphaProteo在生產(chǎn)針對具有挑戰(zhàn)性的目標(如病毒和癌癥相關(guān)蛋白質(zhì))的結(jié)合劑方面表現(xiàn)出色,在許多情況下實現(xiàn)了亞納摩爾親和力。它能夠簡化粘合劑設(shè)計,減少優(yōu)化次數(shù),為治療開發(fā)開辟了新的可能性。
AlphaProteo 在測試的七種目標蛋白質(zhì)上都有較高的實驗成功率。在濕實驗室測試中,9% 到 88% 的候選分子成功結(jié)合,比其他方法高 5 到 100 倍;比現(xiàn)有最佳方法的結(jié)合親和力高 3 到 300 倍。
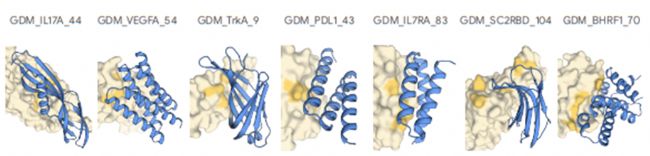
從AI蛋白設(shè)計到濕實驗驗證
研究人員現(xiàn)在能夠直接從序列和結(jié)構(gòu)數(shù)據(jù)中生成針對特定功能(如結(jié)合親和力、穩(wěn)定性和催化活性)優(yōu)化的蛋白質(zhì)。然而,將這些計算設(shè)計轉(zhuǎn)化為功能性、可靠的蛋白質(zhì)需要嚴格的濕實驗驗證,以確保結(jié)合親和力、穩(wěn)定性和生物活性等特性。在濕實驗驗證過程中,科學(xué)家面臨許多挑戰(zhàn),復(fù)雜蛋白質(zhì)的結(jié)構(gòu)和性質(zhì)可能導(dǎo)致其在表達過程中出現(xiàn)折疊異常或形成無活性的聚集體,極大地降低表達效率。濕實驗的數(shù)據(jù)反饋至關(guān)重要,可用于進一步優(yōu)化AI模型,從而提高模型對未來設(shè)計的精準度和有效性。
AI在蛋白質(zhì)科學(xué)中的應(yīng)用多樣性已經(jīng)在重塑藥物發(fā)現(xiàn)、精準醫(yī)學(xué)和合成生物學(xué)。由David Baker等先驅(qū)孵化的公司正在使用深度學(xué)習(xí)來解決關(guān)鍵的生物學(xué)挑戰(zhàn),包括藥物開發(fā)、多肽設(shè)計、小分子結(jié)合蛋白質(zhì)工程和新型材料合成。隨著AI加速和增強蛋白質(zhì)設(shè)計的能力得到證明,其在生物技術(shù)中的作用預(yù)計將不斷增長,為定制療法和創(chuàng)新生物材料開辟新的可能性,并推動生命科學(xué)領(lǐng)域的邊界不斷拓展。
References
[1] Jumper, John, et al. "Highly accurate protein structure prediction with AlphaFold." nature 596.7873 (2021): 583-589.
[2] Baek, Minkyung, et al. "Accurate prediction of protein structures and interactions using a three-track neural network." Science 373.6557 (2021): 871-876.
[3] Madani, Ali, et al. "Large language models generate functional protein sequences across diverse families." Nature Biotechnology 41.8 (2023): 1099-1106.
[4] Senior, Andrew W., et al. "Improved protein structure prediction using potentials from deep learning." Nature 577.7792 (2020): 706-710.
[5] Zambaldi, Vinicius, et al. "De novo design of high-affinity protein binders with AlphaProteo." arXiv preprint arXiv:2409.08022 (2024).
[6] Kulmanov, Maxat, et al. "Protein function prediction as approximate semantic entailment." Nature Machine Intelligence 6.2 (2024): 220-228.
[7] Watson, Joseph L., et al. "De novo design of protein structure and function with RFdiffusion." Nature 620.7976 (2023): 1089-1100.
- AI人工智能在蛋白質(zhì)結(jié)構(gòu)預(yù)測、功能預(yù)測及設(shè)計中的作用
- m5C MeRIP-seq等揭示m5C修飾在癌癥耐藥中的關(guān)鍵調(diào)控機制中的應(yīng)用
- ChIP技術(shù)揭示NURR1在前列腺癌從基因轉(zhuǎn)錄到腫瘤進展中的調(diào)控機制
- 兩種轉(zhuǎn)錄因子在介導(dǎo)番茄果實成熟的表觀遺傳調(diào)控中發(fā)揮的關(guān)鍵作用
- ChIP-seq等在揭示植物H3K27me3相關(guān)轉(zhuǎn)座元件沉默模式中的應(yīng)用
- 文獻解讀:利用WGBS揭示跨代遺傳的DNA甲基化機制
- 合成生物學(xué)之底盤細胞改造對產(chǎn)品開發(fā)的促進作用及優(yōu)勢
- ChIP-seq等技術(shù)揭示突變在癌癥中的H3K36甲基化新型表觀遺傳治療靶點





